library(readxl)
library(janitor)
library(tidyverse)4 Làm sạch (p2)
Bước đầu tiên sau khi đọc được dữ liệu vào R là “làm sạch” dữ liệu (clean data). Chúng ta chỉ tiến hành phân tích khi chắc chắn dữ liệu đã “sạch” và chính xác.
Hiểu khái niệm dữ liệu gọn gàng
Hiểu cách sử dụng các lệnh join
Học các cách xử lý dữ liệu lỗi
4.1 Tổng quan quá trình làm sạch dữ liệu
Quá trình làm sach dữ liệu thường bao gồm các bước theo thứ tự sau
Làm sạch tên cột (Từ phần 1: Section 3.5.1)
Kiểm tra datatype (Từ phần 1: Section 3.4.2)
Kiểm tra giá trị
Format bảng theo quy tắc dữ liệu gọn gàng
Nối bảng (nếu cần thiết)
Xử lý các dữ liệu lỗi
4.2 Làm sạch tên cột & kiểm tra datatype
Chúng ta sẽ dùng vaccine_data.xlsx từ phần trước
df <- read_excel(path = "data/vaccine_data.xlsx", sheet = 1)Như đã được giới thiệu tại phần 1 (Section 3.5.1), các tên cột nên được làm sạch theo các quy tắc của R để tránh gặp lỗi khi chỉnh sửa, trích xuất dữ liệu cột.
Các datatype cũng cần được kiểm tra và update vì mỗi datatype trong R sẽ có các câu lệnh tính toán, phân tích, xử lý khác nhau
Ví dụ:
Lệnh
difftime()dùng để tính khoảng thời gian giữa 2 mốc thời gian yêu cầu 2 tham số phải theo datatype là DateLệnh
sum()dùng để tính tổng của một dãy số yêu cầu tham số phải là 1 vector numeric
Dữ liệu không theo đúng datatype thường sẽ gặp ERROR trong R, nhưng cũng có các trường hợp không có ERROR nhưng trả về kết quả không mong muốn.
# ---- Sử dụng difftime nhưng không chuyển datatype thành Date difftime("05/01/2022", "03/01/2022")Time difference of 731 days# ---- So sánh ngày nhưng không chuyển datatype sang Date print("25/01/2021" < "03/01/2022")[1] FALSE# ---- Tính tổng nhưng không chuyển datatype sang numeric sum(c("1", "3", "4"))Error in sum(c("1", "3", "4")): invalid 'type' (character) of argument
Thực hiện làm sạch tên cột và chỉnh datatype cho dữ liệu vaccine
Dữ liệu trước khi sửa
head(df)Làm sạch tên cột
df <- df %>% clean_names() %>%
rename(
vgb_truoc_24 = vgb_24,
vgb_sau_24 = vgb_24_2
)Code tương ứng khi không dùng dấu %>%
# ---Code không dùng %>%
df <- clean_names(df)
df <- rename(df,
vgb_truoc_24 = vgb_24,
vgb_sau_24 = vgb_24_2
)Kiểm tra và thay đổi datatype
Như đã được giới thiệu trong phần Section 3.4.2, cần chuyển đổi các cột ngày, cột có giá trị phân loại, giá trị TRUE/FALSE sang đúng datatype để thể hiện rõ hơn ý nghĩa giá trị từng cột cũng như tránh xảy ra lỗi khi sử dụng các lệnh xử lý dữ liệu trong R.
# --- Điều chỉnh datatype
date_cols <- c("ngaysinh", "vgb_truoc_24","vgb_sau_24","vgb_1","vgb_2","vgb_3","vgb_4","hg_1","hg_2","hg_3","hg_4","uv_1","uv_2","uv_3","uv_4")
df <- mutate(df,
gioitinh = as.factor(gioitinh),
tinhtrang = ifelse(tinhtrang == "theo dõi", TRUE, FALSE)
) %>%
mutate_at(date_cols, dmy) %>%
rename(theodoi = tinhtrang)Code tương ứng khi không dùng dấu %>%
date_cols <- c("ngaysinh", "vgb_truoc_24","vgb_sau_24","vgb_1","vgb_2","vgb_3","vgb_4","hg_1","hg_2","hg_3","hg_4","uv_1","uv_2","uv_3","uv_4")
df <- mutate(df,
gioitinh = as.factor(gioitinh),
tinhtrang = ifelse(tinhtrang == "theo dõi", TRUE, FALSE)
)
df <- mutate_at(df, date_cols, dmy)
df <- rename(df, theodoi = tinhtrang)Dữ liệu sau khi sửa
head(df)4.3 Kiểm tra giá trị
Thường bao gồm các thao tác chính
Kiểm tra các giá trị NA
Kiểm tra các giá trị bất thường (outlier) và các khoảng giá trị của từng cột
Kiểm tra giá trị giữa các cột (nếu cần thiết)
Trong R có lệnh summary để người dùng có thể nhanh chóng kiểm tra các khoảng giá trị và số dữ liệu NA trong từng cột.
summary(df) id gioitinh ngaysinh huyen
Min. : 1.0 nam:514 Min. :2024-01-01 Length:1000
1st Qu.: 250.8 nữ :486 1st Qu.:2024-01-25 Class :character
Median : 500.5 Median :2024-02-16 Mode :character
Mean : 500.5 Mean :2024-02-16
3rd Qu.: 750.2 3rd Qu.:2024-03-10
Max. :1000.0 Max. :2024-04-01
xa tinh vgb_truoc_24
Length:1000 Length:1000 Min. :0001-01-01
Class :character Class :character 1st Qu.:2024-01-25
Mode :character Mode :character Median :2024-02-17
Mean :2005-10-03
3rd Qu.:2024-03-11
Max. :2024-04-02
NA's :9
vgb_sau_24 vgb_1 vgb_2
Min. :0001-01-01 Min. :0001-01-01 Min. :0001-01-01
1st Qu.:2024-01-26 1st Qu.:2024-02-05 1st Qu.:2024-02-16
Median :2024-02-17 Median :2024-02-27 Median :2024-03-09
Mean :2003-10-11 Mean :2014-01-01 Mean :2009-11-18
3rd Qu.:2024-03-11 3rd Qu.:2024-03-22 3rd Qu.:2024-03-31
Max. :2024-04-03 Max. :2024-04-18 Max. :2024-05-03
NA's :6 NA's :4 NA's :10
vgb_3 vgb_4 hg_1
Min. :0001-01-01 Min. :0001-01-01 Min. :0001-01-01
1st Qu.:2024-02-26 1st Qu.:2024-03-07 1st Qu.:2024-01-26
Median :2024-03-19 Median :2024-03-29 Median :2024-02-17
Mean :2011-12-14 Mean :2012-01-12 Mean :2007-11-11
3rd Qu.:2024-04-10 3rd Qu.:2024-04-21 3rd Qu.:2024-03-11
Max. :2024-05-18 Max. :2024-06-02 Max. :2024-04-02
NA's :10 NA's :6 NA's :5
hg_2 hg_3 hg_4
Min. :0001-01-01 Min. :0001-01-01 Min. :0001-01-01
1st Qu.:2024-02-05 1st Qu.:2024-02-16 1st Qu.:2024-02-26
Median :2024-02-27 Median :2024-03-09 Median :2024-03-18
Mean :1999-09-25 Mean :2009-12-08 Mean :2016-01-24
3rd Qu.:2024-03-21 3rd Qu.:2024-03-31 3rd Qu.:2024-04-10
Max. :2024-04-17 Max. :2024-05-01 Max. :2024-05-16
NA's :6 NA's :6 NA's :7
uv_1 uv_2 uv_3
Min. :0001-01-01 Min. :0001-01-01 Min. :0001-01-01
1st Qu.:2024-01-26 1st Qu.:2024-02-04 1st Qu.:2024-02-15
Median :2024-02-16 Median :2024-02-27 Median :2024-03-09
Mean :2017-12-26 Mean :2013-12-08 Mean :2009-12-08
3rd Qu.:2024-03-11 3rd Qu.:2024-03-21 3rd Qu.:2024-03-31
Max. :2024-04-02 Max. :2024-04-17 Max. :2024-05-02
NA's :12 NA's :10 NA's :6
uv_4 theodoi
Min. :0001-01-01 Mode :logical
1st Qu.:2024-02-26 FALSE:504
Median :2024-03-19 TRUE :496
Mean :2009-12-24
3rd Qu.:2024-04-10
Max. :2024-05-17
NA's :5
Thông tin về các output của lệnh summary
Đối với dữ liệu
numeric:summarysẽ cho các thông tin sauOuput Ý nghĩa Công dụng Min Giá trị nhỏ nhất của cột cùng với Max, kiểm tra khoảng giá trị có hợp lệ không.
VD: đối với cột tuổi,Maxlà 150 có khả năng cao là giá trị saiMax Giá trị lớn nhất của cột cùng với Min, kiểm tra khoảng giá trị có hợp lệ khôngMean Giá trị trung bình của cột Median Số trung vị – giá trị lớn hơn 50% số giá trị trong cột 1st Qu. Tứ phân vị thứ nhất (trung vị phần dưới) – giá trị lớn hơn 25% số giá trị trong cột cung cấp thêm thông tin về khoảng giá trị của cột, có thể được sử dụng để tính outlier 3rd Qu. Tứ phân vị thứ 3 (trung vị phần trên) - giá trị lớn hơn 75% số giá trị trong cột cung cấp thêm thông tin về khoảng giá trị của cột, có thể được sử dụng để tính outlier NA Số giá trị trống trong cột Đối với dữ liệu
character:summarycho biết tổng số hàng và số dữ liệuNAĐối với dữ liệu
factorhaylogical:summarysẽ cho biết số lượng dữ liệu cho từng nhóm và số dữ liệuNA
4.3.1 Giá trị NA
Giá trị NA (hay giá trị missing) là các giá trị được bỏ trống
Trong R, NA là một giá trị đặc biệt khi không thể tính toán hay so sánh với các datatype khác (kết quả luôn trả về NA).
Vì vậy dữ liệu NA nên được xử lý trước khi thực hiện tính toán hay phân tích với dữ liệu.
Ví dụ:
# các phép tính toán khi có NA sẽ trả về NA
sum(c(1, 2, 3, 4, NA))[1] NA# khi thống kê với dữ liệu có chứa NA sẽ trả về NA
max(c(1, 2, 3, 4, NA))[1] NAXử lý giá trị NA
Tuỳ vào trường hợp, các giá trị NA có thể được xử lý 1 trong 2 cách
Thay thế NA bằng một giá trị khác (Section 4.6.1)
Loại bỏ các hàng chứa
NA(Section 4.6.2)
4.3.2 Outlier
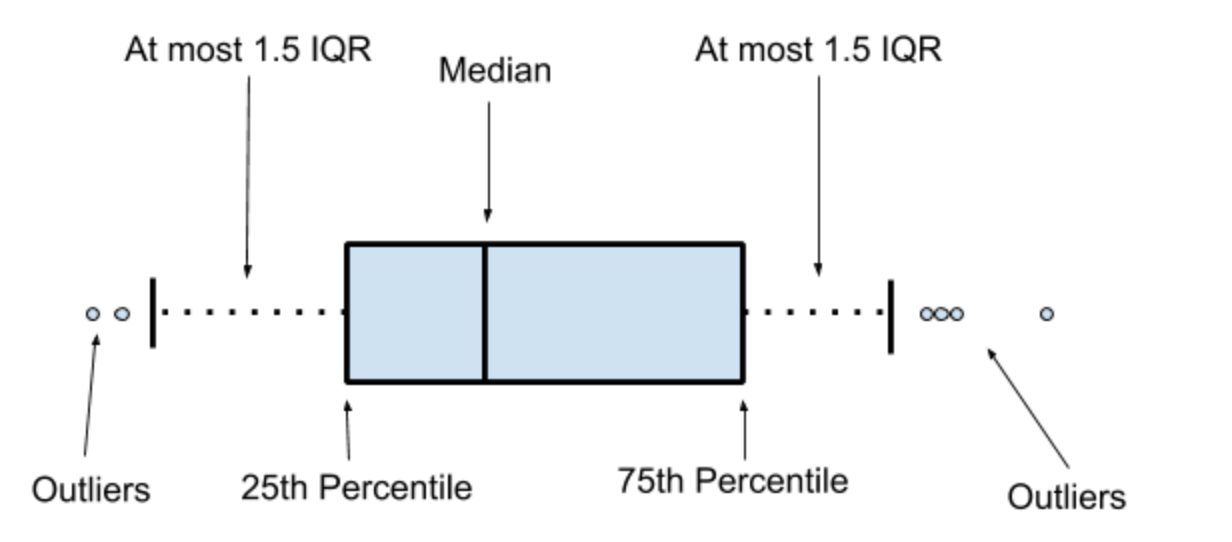
Outlier là các giá trị khác đáng kể so với các giá trị khác
Cách xác định outlier đơn giản nhất là bằng cách sử dụng thuật toán Tukey. Trong đó, outlier được định nghĩa là:
Các giá trị lớn hơn
Q3 + (Q3 - Q1)*1.5Các giá trị nhỏ hơn
Q1 - (Q3 - Q1)*1.5
Về mặt quản lý dữ liệu, outlier thường là dấu hiệu cho thấy dữ liệu được nhập có thể là dữ liệu sai cần được kiểm tra lại
Về mặt phân tích, outlier có thể gây ra độ lệch rất lớn trong một kết quả thống kê
Ví dụ:
# ---- ví dụ dãy số có outlier
example_outlier <- c(-10, 1,3,4,1,2,5,20)
# --- kết quả tính mean khi có outlier
mean(example_outlier)[1] 3.25# --- kết quả tính mean sau khi lọc outlier theo công thức xác định outlier
q1 <- quantile(example_outlier, 0.25)
q3 <- quantile(example_outlier, 0.75)
iqr <- q3 - q1
filter_outlier <- subset(example_outlier, (example_outlier > q1 - iqr*1.5)&(example_outlier < q3 + iqr*1.5))
mean(filter_outlier)[1] 2.666667Để nhanh chóng kiểm tra dữ liệu có chứa outlier hay không, ta có thể sử dụng boxplot để tìm các điểm giá trị cách xa các giá trị còn lại
Ví dụ: sử dụng boxplot để kiểm tra outlier của cột vgb_1
boxplot(df$vgb_1)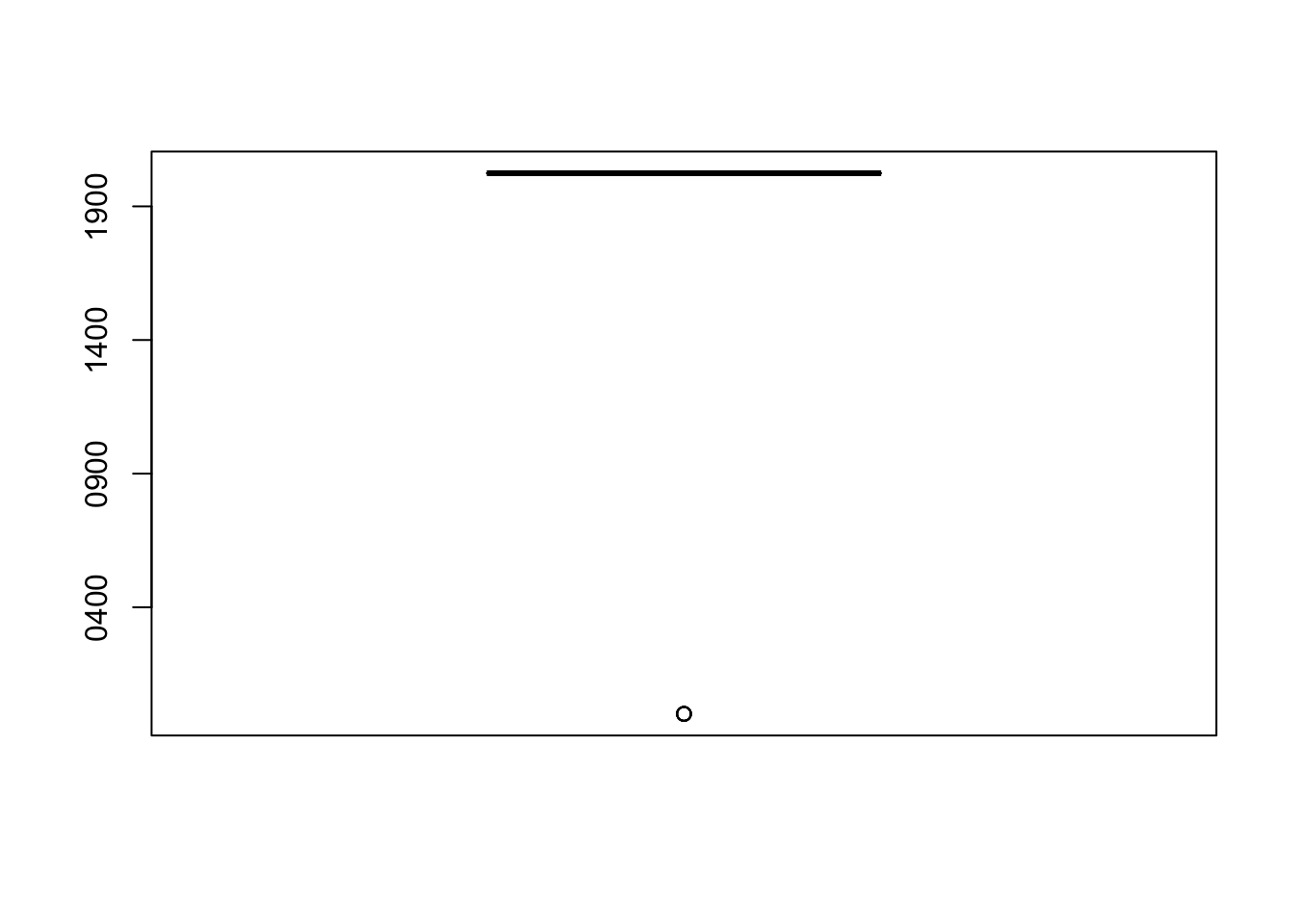
# xem các giá trị được phân loại là outlier
boxplot(df$vgb_1, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"Thực hiện phép kiểm tra outlier trên các cột còn lại của dữ liệu vaccine
# --- các mũi vgb
boxplot(df$vgb_truoc_24, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"boxplot(df$vgb_sau_24, plot=FALSE)$out [1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"boxplot(df$vgb_2, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01"boxplot(df$vgb_3, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01"boxplot(df$vgb_4, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01"# ---- hg
boxplot(df$hg_1, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01" "0001-01-01"boxplot(df$hg_2, plot=FALSE)$out [1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[11] "0001-01-01" "0001-01-01"boxplot(df$hg_3, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01"boxplot(df$hg_4, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"# --- uv
boxplot(df$uv_1, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01"boxplot(df$uv_2, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"boxplot(df$uv_3, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01"boxplot(df$uv_4, plot=FALSE)$out[1] "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01" "0001-01-01"
[6] "0001-01-01" "0001-01-01"Xử lý outlier
Trong nhiều trường hợp, các dữ liệu được xem là outlier vẫn có thể là một giá trị hợp lệ và nên được giữ cho việc phân tích.
Tuy nhiên, nếu outlier được xác định là dữ liệu sai (trong bộ dữ liệu vaccine, ngày tiêm “01-01-0001” có thể được xác định là sai) thì có thể xem các outlier đó là dữ liệu NA và xử lý một cách tương tự
4.3.3 Kiểm tra giá trị giữa các cột
Ngoài ra, tuỳ vào bộ dữ liệu, có thể cần kiểm tra giá trị giữa các cột (VD: trong dữ liệu tiêm chủng có thể có mũi 2 trùng hoặc trước ngày tiêm mũi 1)
Kiểm tra các hàng có ngày tiêm không đúng thứ tự
# --- Kiểm tra các mũi vgb
incorrect_vgb <- df %>%
filter(
(vgb_truoc_24 > vgb_sau_24) | (vgb_sau_24 > vgb_1) | (vgb_1 > vgb_2) | (vgb_2 > vgb_3) | (vgb_3 > vgb_4)
) %>%
select(vgb_truoc_24, vgb_sau_24, vgb_1, vgb_2, vgb_3, vgb_4)
incorrect_vgb# --- Kiểm tra các mũi ho gà
incorrect_hg <- df %>%
filter(
(hg_1 > hg_2) | (hg_2 > hg_3) | (hg_3 > hg_4)) %>%
select(hg_1, hg_2, hg_3, hg_4)
incorrect_hg# --- Kiểm tra các mũi uốn ván
incorrect_uv <- df %>%
filter(
(uv_1 > uv_2) | (uv_2 > uv_3) | (uv_3 > uv_4) )%>%
select(uv_1, uv_2, uv_3, uv_4)
incorrect_uv4.4 Dữ liệu gọn gàng (tidy data)
4.4.1 Dữ liệu gọn gàng là gì?
Dữ liệu gọn gàng cần có các đặc điểm sau:1
- Mỗi biến là một cột; mỗi cột là một biến
- Mỗi quan sát là một hàng; mỗi hàng là một quan sát
- Mỗi giá trị nằm trong một ô; mỗi ô chứa một giá trị duy nhất
Ví dụ: dữ liệu không gọn gàng và gọn gàng
Dữ liệu không gọn gàng
| country | code | 2015 | 2016 |
|---|---|---|---|
| Aruba | ABW | 28419.2645 | 28449.713 |
| Africa Eastern and Southern | AFE | 1554.1673 | 1444.004 |
| Afghanistan | AFG | 566.8811 | 523.053 |
| Africa Western and Central | AFW | 1882.2640 | 1648.763 |
| Angola | AGO | 3217.3392 | 1809.709 |
| Albania | ALB | 3952.8036 | 4124.055 |
Bảng ví dụ này đang thể hiện GDP của từng quốc gia qua từng năm. Trong đó, chủ thể sẽ là các quốc gia và các đặc điểm là năm và GDP
Đối chiếu với các đặc điểm của tidy data nêu trên, bảng dữ liệu này không phải là dữ liệu gọn gàng vì những lí do sau:
2015, 2016 không phải là biến vì chúng không phải là các đặc điểm của chủ thể (trong VD này là các quốc gia), mà là giá trị cho biến (đặc điểm) năm
Trong bảng dữ liệu này, mỗi quan sát sẽ là GDP của 1 quốc gia trong 1 năm, vì vậy bảng dữ liệu này cũng đang vi phạm quy tắc của tidy data vì mỗi hàng đang chứa 2 quan sát (GDP cho 2 năm)
Cách sắp xếp hiện tại còn được gọi là dữ liệu dạng ngang (wide) vì các quan sát cho một chủ thể đều được sắp xếp trong 1 hàng.
Dữ liệu gọn gàng
Để bảng ví dụ GDP tuân theo quy tắc tidy, ta cần tạo một cột mới cho biến năm và cột gdp để chứa giá trị GDP tương ứng.
Bảng dữ liệu GDP theo quy tắc tidy data sẽ như sau
| country | code | year | gdp |
|---|---|---|---|
| Aruba | ABW | 2015 | 28419.2645 |
| Aruba | ABW | 2016 | 28449.7129 |
| Africa Eastern and Southern | AFE | 2015 | 1554.1673 |
| Africa Eastern and Southern | AFE | 2016 | 1444.0035 |
| Afghanistan | AFG | 2015 | 566.8811 |
| Afghanistan | AFG | 2016 | 523.0530 |
| Africa Western and Central | AFW | 2015 | 1882.2640 |
| Africa Western and Central | AFW | 2016 | 1648.7627 |
| Angola | AGO | 2015 | 3217.3392 |
| Angola | AGO | 2016 | 1809.7094 |
| Albania | ALB | 2015 | 3952.8036 |
| Albania | ALB | 2016 | 4124.0554 |
Cách sắp xếp hiện tại còn được gọi là dữ liệu định dạng dọc (long) vì các quan sát được xếp theo chiều dọc (mỗi hàng chỉ chứa 1 quan sát duy nhất).
4.4.2 Format bảng dữ liệu vaccine
Dữ liệu trước khi format
head(df, n = 10)Bảng dữ liệu vaccine đang thể hiện thông tin về từng đợt tiêm vaccine của từng trẻ. Vì vậy, các biến sẽ là các đặc điểm của 1 đợt tiêm, và mỗi quan sát sẽ là thông tin về mỗi đợt tiêm (có thể hiểu là giá trị của các biến cho mỗi đợt tiêm khác nhau)
Bảng dữ liệu hiện tại chưa tuân theo quy tắc tidy data vì các lý do sau:
- Mỗi hàng đang chứa nhiều quan sát (chứa “ngày tiêm” của nhiều lần tiêm khác nhau).
- Các cột kháng nguyên (
vgb_1,vgb_2, …) là giá trị cho đặc điểm “loại kháng nguyên” của các đợt tiêm khác nhau.
Để tuân theo quy tắc tidy data, ta cần format bảng dữ liệu sang định dạng dọc (long)
Để format bảng dữ liệu trong R, ta có thể sử dụng các lệnh sau.
pivot_longer()- chuyển dữ liệu từ dạng ngang sang dọc.pivot_wider()- chuyển dữ liệu từ dạng dọc sang ngang.
cols <- which(str_ends(colnames(df), "[:digit:]"))
colnames(df) [1] "id" "gioitinh" "ngaysinh" "huyen" "xa"
[6] "tinh" "vgb_truoc_24" "vgb_sau_24" "vgb_1" "vgb_2"
[11] "vgb_3" "vgb_4" "hg_1" "hg_2" "hg_3"
[16] "hg_4" "uv_1" "uv_2" "uv_3" "uv_4"
[21] "theodoi" str_ends(colnames(df), "[:digit:]") [1] FALSE FALSE FALSE FALSE FALSE FALSE TRUE TRUE TRUE TRUE TRUE TRUE
[13] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE FALSEcolnames(df)[cols] [1] "vgb_truoc_24" "vgb_sau_24" "vgb_1" "vgb_2" "vgb_3"
[6] "vgb_4" "hg_1" "hg_2" "hg_3" "hg_4"
[11] "uv_1" "uv_2" "uv_3" "uv_4" df <- pivot_longer(df,
cols = any_of(cols),
# đặt tên mới cho biến
names_to = "khangnguyen",
# đặt tên mới cho cột giá trị tương ứng
values_to = "ngaytiem") Dữ liệu sau khi format
head(df, n=10)4.5 Nối bảng
Trong nhiều trường hợp, dữ liệu bị chia thành nhiều bảng khác nhau và để phân tích thì chúng ta phải nối các bảng lại. (Lưu ý: để nối 2 bảng với nhau, 2 bảng cần có các cột chứa giá trị để xác định các hàng tương ứng cần được nối)
Ví dụ: Có 2 bảng bệnh nhân và bệnh viện như sau
Bảng bệnh nhân
| patient_id | age | gender | hospital_id |
|---|---|---|---|
| 1 | 28 | nam | 1 |
| 2 | 30 | nữ | 2 |
| 3 | 10 | nam | 1 |
| 4 | 12 | nữ | 1 |
Bảng bệnh viện
| id | hospital_name | address |
|---|---|---|
| 1 | Bệnh viện Bệnh Nhiệt đới | 764 Đ. Võ Văn Kiệt, Phường 1, Quận 5 |
| 2 | Bệnh viện Nhi đồng 1 | 341 Đ. Sư Vạn Hạnh, Phường 10, Quận 10 |
Để có đầy đủ thông tin bệnh nhân và bệnh viện, ta cần ghép 2 bảng với nhau. Cột để xác định các hàng cần được nối là cột hospital_id bên bảng bệnh nhân và cột id bên bảng bệnh viện.
Bảng sau khi nối sẽ như sau
| patient_id | age | gender | hospital_id | hospital_name | address |
|---|---|---|---|---|---|
| 1 | 28 | nam | 1 | Bệnh viện Bệnh Nhiệt đới | 764 Đ. Võ Văn Kiệt, Phường 1, Quận 5 |
| 2 | 30 | nữ | 2 | Bệnh viện Nhi đồng 1 | 341 Đ. Sư Vạn Hạnh, Phường 10, Quận 10 |
| 3 | 10 | nam | 1 | Bệnh viện Bệnh Nhiệt đới | 764 Đ. Võ Văn Kiệt, Phường 1, Quận 5 |
| 4 | 12 | nữ | 1 | Bệnh viện Bệnh Nhiệt đới | 764 Đ. Võ Văn Kiệt, Phường 1, Quận 5 |
4.5.1 Nối bảng trong R
Để nối 2 bảng với nhau trong R, ta sử dụng 1 trong các lệnh join của package dplyr sau đây
| Function | Công dụng | GIF minh hoạ |
|---|---|---|
| left_join() | Nối ngoài bên trái: lấy bảng bên trái làm chuẩn, và tìm kiếm các hàng tương ứng của bảng phải để nối. Nếu không có hàng tương ứng để nối thì sẽ để giá trị |
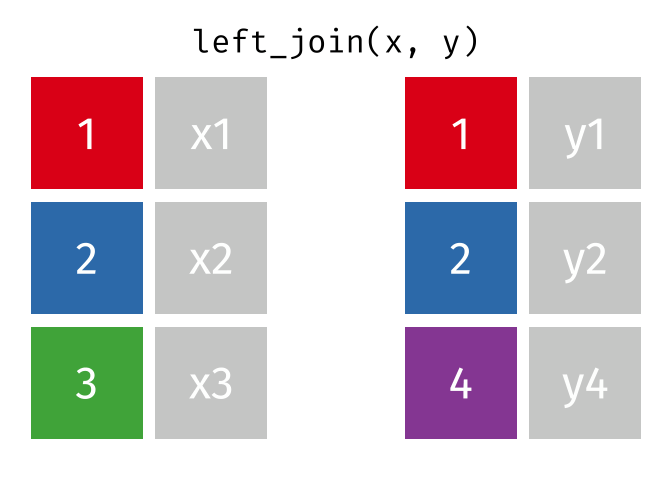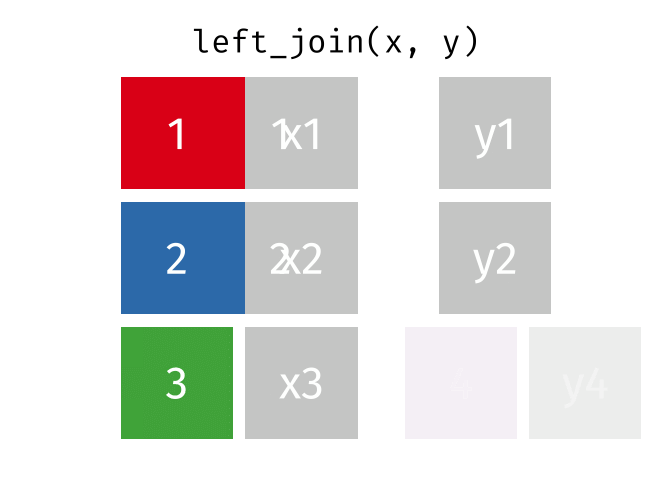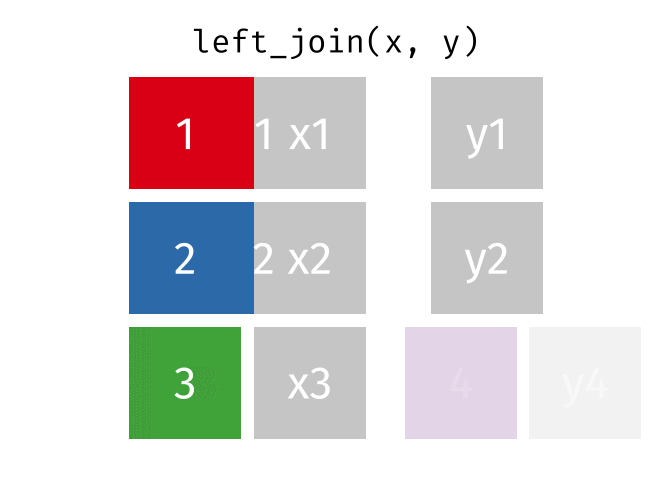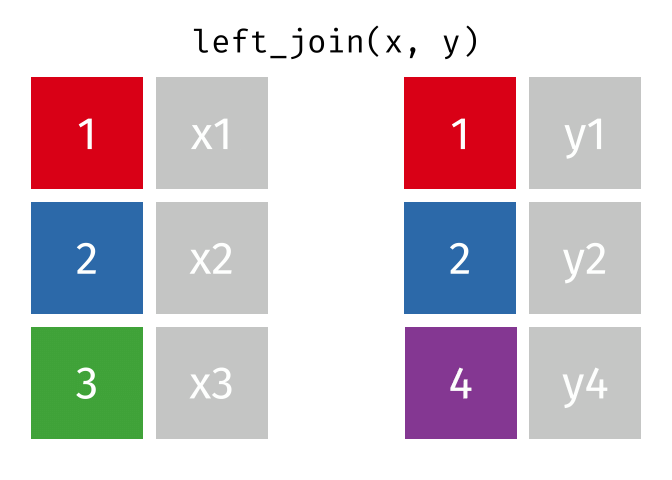 |
| inner_join() | Nối trong: lấy bảng bên trái làm chuẩn, và tìm kiếm các hàng tương ứng của bảng phải để nối. Các hàng không có hàng tương ứng để nối sẽ bị xoá |
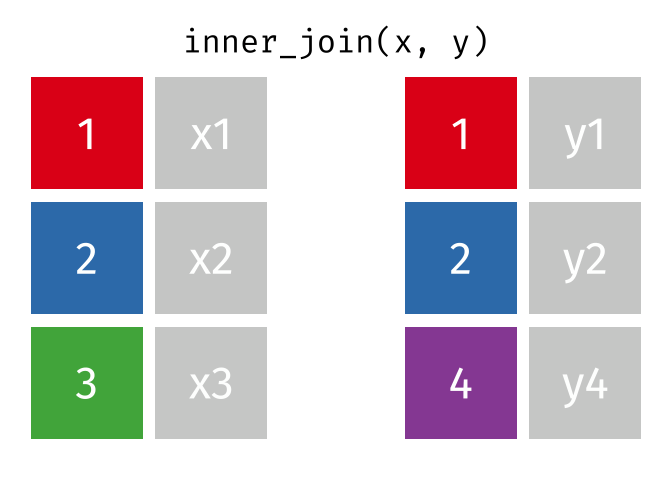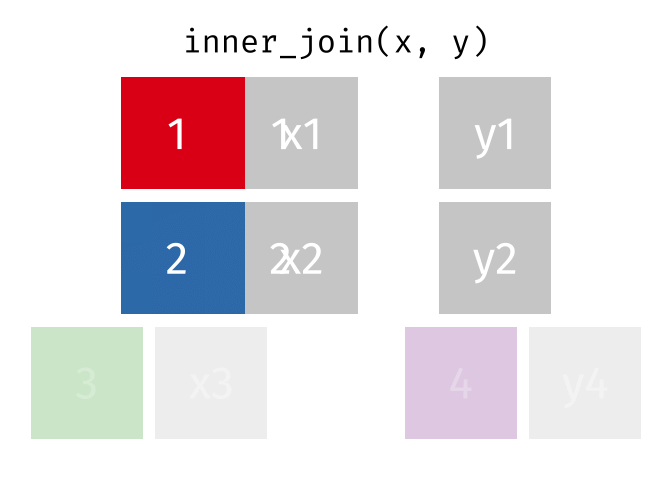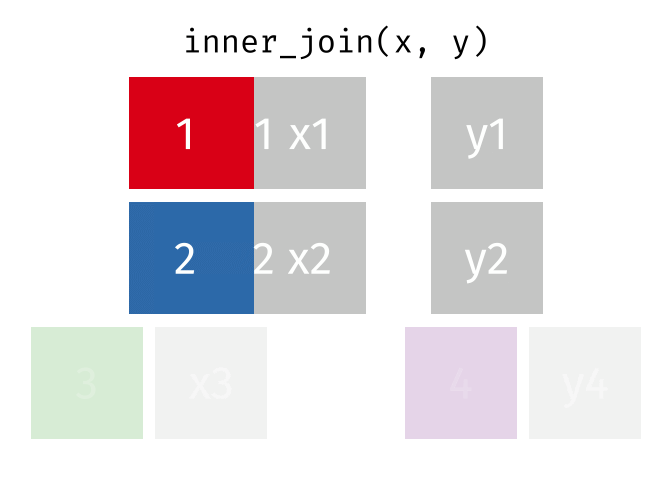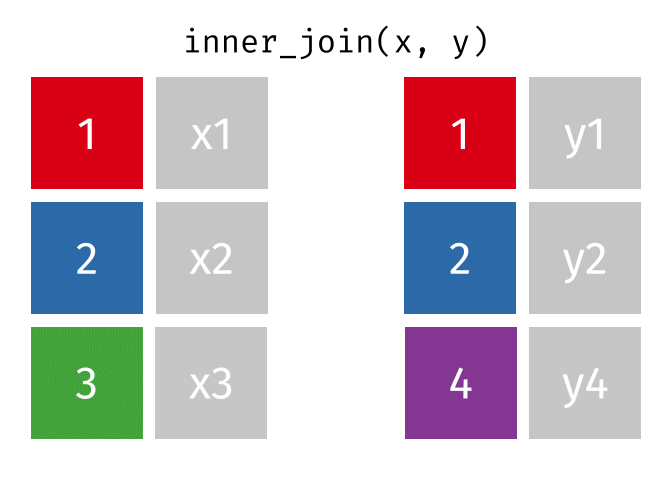 |
| full_join() | Nối ngoài: giữ tất cả các hàng của 2 bảng sau khi nối Các hàng của 2 bảng không nối được sẽ được chèn giá trị |
 |
Để sử dụng lệnh join, ta cần cung cấp cho R các thông tin sau:
2 bảng cần nối
các cột chung để nối bằng lệnh
join_by(R mặc định nối 2 bảng bằng cột có tên giống nhau)
Tổng hợp các lệnh join cùng GIF minh hoạ
Nối bảng dữ liệu vaccine
Thực hiện nối bảng trên dữ liệu vaccine để kiểm tra và loại bỏ các hàng quận/huyện, phường/xã không hợp lệ
Thông tin về bảng dữ liệu qhpx_hcm.rds
Dùng để đối chiếu và kiểm tra xem các quận huyện trong bộ dữ liệu vaccine có hợp lệ hay không (quận + huyện có tồn tại hay không). Ví dụ một số dữ liệu không hợp lệ [ (Quận 6, Xã Bình Trung), (Quận 6, Xã Xuân Bắc), …]
Đồng thời cung cấp tên quận và xã được đồng nhất
Từ điển biến số
| Tên biến | Ý nghĩa |
|---|---|
| qh | tên quận/huyện xuất hiện trong bộ dữ liệu vaccine |
| px | tên phường/xã xuất hiện trong bộ dữ liệu vaccine |
| qh_update | tên quận/huyện được đồng bộ hoá theo format “Quận số” đối với các quận có tên số hoặc “Tên quận” đối với quận chữ VD: “Quận Thủ Đức”/ “Thành phố Thủ đức” được đồng bộ hoá thành “Thủ Đức” |
| px_update | tên phường/xã được đồng bộ hoá theo format “Phường số” đối với các phường có tên số hoặc “Tên phường” đối với phường chữ |
| ma_px | mã cho phường/xã |
# --- Đọc bảng quận huyện phường xã
qhpx <- readRDS("data/qhpx_hcm.rds")
qhpxBảng qhpx
head(qhpx, n=10)Bảng dữ liệu vaccine
head(df, n=10)2 bảng sẽ được nối bằng giá trị cột huyện và xã
# --- Nối 2 bảng bằng lệnh left_join
joined_df <- left_join(df, qhpx, by = join_by(huyen == qh, xa == px)) Sau khi nối bằng left_join, các hàng của bảng trái (df) không nối được với bảng phải (qhpx) sẽ được chèn giá trị NA. Vì vậy, các hàng có qh_update hoặc px_update là NA sẽ là các hàng có quận/huyện và phường/xã không khợp lệ
Ta có thể xem các hàng có lỗi như sau
joined_df %>% filter(is.na(qh_update))4.6 Xử lý dữ liệu
Thường bao gồm
Chỉnh sửa giá trị dữ liệu
Lọc trùng
Lọc hàng theo điều kiện
4.6.1 Chỉnh sửa giá trị dữ liệu
Các trường hợp có thể cần chỉnh sửa lại dữ liệu:
Dữ liệu NA (trống)
Các dữ liệu dạng TRUE/FALSE được hiển thị bằng cách khác trong dữ liệu gốc (VD: Đánh dấu X hoặc bỏ trống trong file excel)
Đồng nhất dữ liệu
Một số lệnh liên quan:
replace_nathay thế giá trịNAbằng giá trị được cung cấpis.nachuyển thành định dạng logical, các dữ liệu trống (NA) có giá trị TRUE và ngược lạiifelsemã hoá theo điều kiện đơn giản, chỉ có 2 trường hợp khi đáp ứng điều kiện hoặc không.case_whenmã hóa giá trị cho nhiều trường hợp nhất định
Code mô phỏng sử dụng các lệnh trên
# --- Thay thế các giá trị NA trong cột col1 thành "default"
df %>%
mutate(col1 = replace_na(col1, "default"))
# --- Thay thế các giá trị được đánh dấu trong col2 thành TRUE và ngược lại
df %>%
mutate(col2 = !is.na(col2))
# Cách làm khác bằng ifelse
df %>%
mutate(col2 = ifelse(is.na(col2), FALSE, TRUE))
# --- Mã hoá giá trị cho nhiều trường hợp
df %>%
mutate(col2 = case_when(
condition_1 ~ val_1, # nếu col2 thoả điều kiện condition_1 -> gán giá trị val_1
condition_2 ~ val_2, # nếu col2 thoả điều kiện condition_2 -> gán giá trị val_2
condition_3 ~ val_3, # nếu col2 thoả điều kiện condition_3 -> gán giá trị val_3
.default = default_val # trong các trường hợp khác -> gán giá trị default_val
))Thực hiện các bước chỉnh sửa giá trị dữ liệu trên bộ dữ liệu vaccine
Đồng nhất tên huyện, xã theo cột qh_update, px_update
joined_df <- joined_df %>% mutate(huyen = qh_update, xa = px_update)Xem dữ liệu sau khi sửa
# n = 100 để hiện 100 hàng đầu thay vì toàn bộ bảng dữ liệu
head(joined_df, n =100)Đối với trường hợp này, các giá trị NA sẽ được loại bỏ ở bước tiếp theo thay vì được thay thế bằng các giá trị khác
Optional: Code sửa thứ tự mũi tiêm
df %>%
mutate(
khangnguyen = str_remove_all(khangnguyen, "_.*")
) %>%
unique() %>% # lọc trùng cùng kháng nguyên trong 1 ngày
mutate(
# tính các mũi vgb sơ sinh trước và sau 24h
khangnguyen = case_when(
khangnguyen == "vgb" & difftime(ngaytiem, ngaysinh, units = "days") < 1 ~ "vgb_truoc_24",
khangnguyen == "vgb" & difftime(ngaytiem, ngaysinh, units = "days") == 1 ~ "vgb_sau_24",
.default = khangnguyen
)
) %>%
group_by(
id, khangnguyen
) %>%
arrange(ngaytiem) %>%
mutate(
shot_count = 1:n(), # tính lại thứ tự mũi tiêm
exclude_count = sum( str_detect(khangnguyen, "24") ), # loại các mũi sơ sinh khỏi số tt
shot_count = shot_count - exclude_count, # loại các mũi sơ sinh khỏi số tt
khangnguyen = if_else(str_detect(khangnguyen, "24"), khangnguyen, paste(khangnguyen, shot_count, sep = "_")) # update tên mũi
) %>%
ungroup() %>%
select(-shot_count, -exclude_count) # A tibble: 13,424 × 9
id gioitinh ngaysinh huyen xa tinh theodoi khangnguyen ngaytiem
<dbl> <fct> <date> <chr> <chr> <chr> <lgl> <chr> <date>
1 26 nam 2024-01-03 Quận 10 Phườ… Thàn… FALSE vgb_truoc_… 0001-01-01
2 28 nam 2024-03-31 Thành p… Xã T… Thàn… FALSE vgb_truoc_… 0001-01-01
3 28 nam 2024-03-31 Thành p… Xã T… Thàn… FALSE uv_1 0001-01-01
4 30 nam 2024-01-03 Quận 6 Xã N… Thàn… TRUE hg_1 0001-01-01
5 30 nam 2024-01-03 Quận 6 Xã N… Thàn… TRUE uv_1 0001-01-01
6 60 nam 2024-01-06 Huyện T… Phườ… Thàn… FALSE vgb_truoc_… 0001-01-01
7 60 nam 2024-01-06 Huyện T… Phườ… Thàn… FALSE hg_1 0001-01-01
8 86 nam 2024-02-15 Quận 6 Phườ… Thàn… FALSE vgb_truoc_… 0001-01-01
9 86 nam 2024-02-15 Quận 6 Phườ… Thàn… FALSE uv_1 0001-01-01
10 109 nữ 2024-01-30 Quận 1 Tân … Thàn… FALSE vgb_truoc_… 0001-01-01
# ℹ 13,414 more rows4.6.2 Lọc hàng
Bước làm sạch điển hình sau khi đã làm sạch các cột và các giá trị được mã hóa lại đó là lọc bộ dữ liệu cho các hàng cụ thể
Để lọc hàng, ta dùng lệnh filter kết hợp với điều kiện lọc như đã học ở phần Section 3.5.4
Một số điều kiện lọc:
Loại bỏ
NA:!is.na()Lọc bằng số thứ tự hàng
Lọc theo giá trị
drop_NA
drop_NA là lệnh để nhanh chóng drop các hàng có chứa giá trị NA (ở bất kỳ cột nào)
Nếu tên cột được cung cấp, drop_NA chỉ drop các hàng có giá trị NA ở các cột được quy định
# --- drop tất cả các hàng có giá trị NA
df %>% drop_na()
# --- chỉ drop các hàng có giá trị NA tại cột ngaytiem
df %>% drop_na(ngaytiem)Việc lọc với điều kiện lớn hơn (>) hoặc nhỏ hơn (<) một ngày hoặc số có thể loại bỏ bất kỳ hàng nào có giá trị NA. Điều này là do NA được coi là giá trị lớn hoặc nhỏ vô hạn.
Thực hiện lọc giá trị trên dữ liệu vaccine
Loại bỏ các dữ liệu có qh px không hợp lý
joined_df <- joined_df %>% filter(!is.na(qh_update)) Loại bỏ các dữ liệu có ngày tiêm NA
joined_df <- joined_df %>% drop_na(ngaytiem)Loại bỏ các ngày tiêm trước ngày sinh (trong bộ dữ liệu vaccine này, lệnh filter sau cũng loại bỏ luôn các outlier)
joined_df <- joined_df %>% filter(ngaytiem >= ngaysinh)4.6.3 Lọc trùng
Lệnh distinct của gói dplyr sẽ dữ lại 1 hàng duy nhất trong các hàng có giá trị bị trùng.
Lệnh distinct mặc định sẽ kiểm tra giá trị của tất cả các cột (2 hàng được đánh giá là trùng khi tất cả các trị của chúng giống nhau).
Thực hiện lọc trùng cho bộ dữ liệu vaccine
joined_df <- joined_df %>% distinct()4.6.4 Dữ liệu vaccine sau quá trình xử lý
4.7 Biến đổi cột
4.7.1 Chọn cột
Lệnh select của dplyr thường được sử dụng để chọn lọc cột một cách nhanh chóng
Để chọn cột, chỉ cần gõ tên các cột được chọn, phân cách bởi dấu
,Để xoá cột, thêm dấu
-trước tên cột
Xóa cột không cần thiết trong dữ liệu vaccine
xoá các cột qh_update, px_update sau khi đã chỉnh sửa tên quận, xã
# --- Xóa cột
joined_df <- joined_df %>% select(-qh_update, -px_update)4.7.2 Tạo cột mới
Như đã được nhắc đến trong các phần trước, lệnh mutate được sử dụng để tạo cột mới hoặc chỉnh sửa giá trị của cột.
Ví dụ 1: tạo cột mới tên qhpx kết hợp cột huyen, xa
example_qhpx <- joined_df %>%
mutate(
# tạo cột mới kết hợp quận và phường
qhpx = str_glue("{huyen}_{xa}"),
)
head(example_qhpx, n=10)Ví dụ 2: tạo cột nhóm tuổi với case_when
example_age_group <- joined_df %>%
mutate(
tuoi = difftime(ngaytiem, ngaysinh, units = "days")/365,
nhom_tuoi = case_when(
tuoi < 1 ~ "0 - 1",
tuoi < 10 ~ "1 - 10",
tuoi < 20 ~ "10 - 20",
.default = "> 20"
)
)
head(example_age_group, n=10)4.8 Lưu dữ liệu sau khi xử lý
Sử dụng lệnh saveRDS() từ phần Section 3.8 để lưu dữ liệu đã qua xử lý
saveRDS(joined_df, "cleaned_vacdata.rds")4.9 Bài tập
Đọc dữ liệu từ sheet 1 của file gdp-capita.xlsx và làm sạch tên cột
Thông tin về bộ dữ liệu
Bảng dữ liệu chứa thông tin về GDP của các quốc gia qua các năm (từ 2015 - 2022)
| Tên cột | Ý nghĩa |
|---|---|
| country | tên quốc gia |
| code | mã quốc gia 3 ký tự |
| `2015` `2016` `2017` `2018` `2019` `2020` `2021` `2022` | các cột năm |
Code
library(readxl)
library(janitor)
library(tidyverse)
gdp <- read_excel(path = "data/gdp-capita.xlsx", sheet = 1) %>% clean_names()4.9.1 Format bảng
Điều chỉnh định dạng dữ liệu đọc dược theo quy tắc dữ liệu gọn gàng
Gợi ý
Bảng dữ liệu hiện tại chưa tuân theo quy tắc tidy data vì các cột 2015 - 2022 không phải là biến và mỗi hàng đang chứa nhiều quan sát (quan sát cho các năm từ 2015 - 2022).
Hiện tại bảng dữ liệu đang ở định dạng ngang (wide), khi nhiều quan sát của cùng chủ thể (trong VD này là các quốc gia) được lưu trữ trong một hàng duy nhất.
Code
gdp <- gdp %>%
pivot_longer(
cols = starts_with("x"),
# đặt tên mới cho biến
names_to = "year",
# đặt tên mới cho cột giá trị tương ứng
values_to = "gdp")
head(gdp)# A tibble: 6 × 4
country_name country_code year gdp
<chr> <chr> <chr> <dbl>
1 Aruba ABW x2015 28419.
2 Aruba ABW x2016 28450.
3 Aruba ABW x2017 29329.
4 Aruba ABW x2018 30918.
5 Aruba ABW x2019 31903.
6 Aruba ABW x2020 24008.4.9.2 Nối bảng
Đọc sheet thứ 2 của file gdp-capita.xlsx chứa thông tin từng quốc gia và nối với bảng dữ liệu đã được chỉnh sửa
2 bảng có cột chung là country_code
Code
mt <- read_excel(path = "data/gdp-capita.xlsx", sheet = 2) %>%
clean_names()
# --- Nối 2 bảng bằng lệnh left_join
# Trong trường hợp này, tuy lệnh join_by không cần thiết nhưng có thể giúp cho code rõ ràng hơn
joined_gdp <- left_join(gdp, mt, by = join_by(country_code == country_code))
head(joined_gdp)# A tibble: 6 × 8
country_name country_code year gdp region income_group special_notes
<chr> <chr> <chr> <dbl> <chr> <chr> <chr>
1 Aruba ABW x2015 28419. Latin Ameri… High income <NA>
2 Aruba ABW x2016 28450. Latin Ameri… High income <NA>
3 Aruba ABW x2017 29329. Latin Ameri… High income <NA>
4 Aruba ABW x2018 30918. Latin Ameri… High income <NA>
5 Aruba ABW x2019 31903. Latin Ameri… High income <NA>
6 Aruba ABW x2020 24008. Latin Ameri… High income <NA>
# ℹ 1 more variable: table_name <chr>mt# A tibble: 265 × 5
country_code region income_group special_notes table_name
<chr> <chr> <chr> <chr> <chr>
1 ABW Latin America & Caribbean High income <NA> Aruba
2 AFE <NA> <NA> "26 countrie… Africa Ea…
3 AFG South Asia Low income "The reporti… Afghanist…
4 AFW <NA> <NA> "22 countrie… Africa We…
5 AGO Sub-Saharan Africa Lower middl… "The World B… Angola
6 ALB Europe & Central Asia Upper middl… <NA> Albania
7 AND Europe & Central Asia High income <NA> Andorra
8 ARB <NA> <NA> "Arab World … Arab World
9 ARE Middle East & North Africa High income <NA> United Ar…
10 ARG Latin America & Caribbean Upper middl… "The World B… Argentina
# ℹ 255 more rows4.9.3 Tạo cột mới
Tạo cột mới tên country_details nối cột country_name và country_code theo format "Name: [country_name] Code: [country_code]"
Code
joined_gdp %>%
mutate(
# tạo cột mới kết hợp 3 cột country_name, country_code, region
country_details = str_glue("Name: {country_name} Code:{country_code} Region: {region}"),
.keep = "used", # giữ các cột được sử dụng
.before = country_name # đặt cột country_details trước cột country_name
)# A tibble: 2,128 × 4
country_details country_name country_code region
<glue> <chr> <chr> <chr>
1 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
2 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
3 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
4 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
5 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
6 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
7 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
8 Name: Aruba Code:ABW Region: Latin America … Aruba ABW Latin…
9 Name: Africa Eastern and Southern Code:AFE … Africa East… AFE <NA>
10 Name: Africa Eastern and Southern Code:AFE … Africa East… AFE <NA>
# ℹ 2,118 more rows