library(dplyr)
library(tidyr)
library(ggplot2)6 Trực quan hóa 1
6.1 Biểu đồ 1
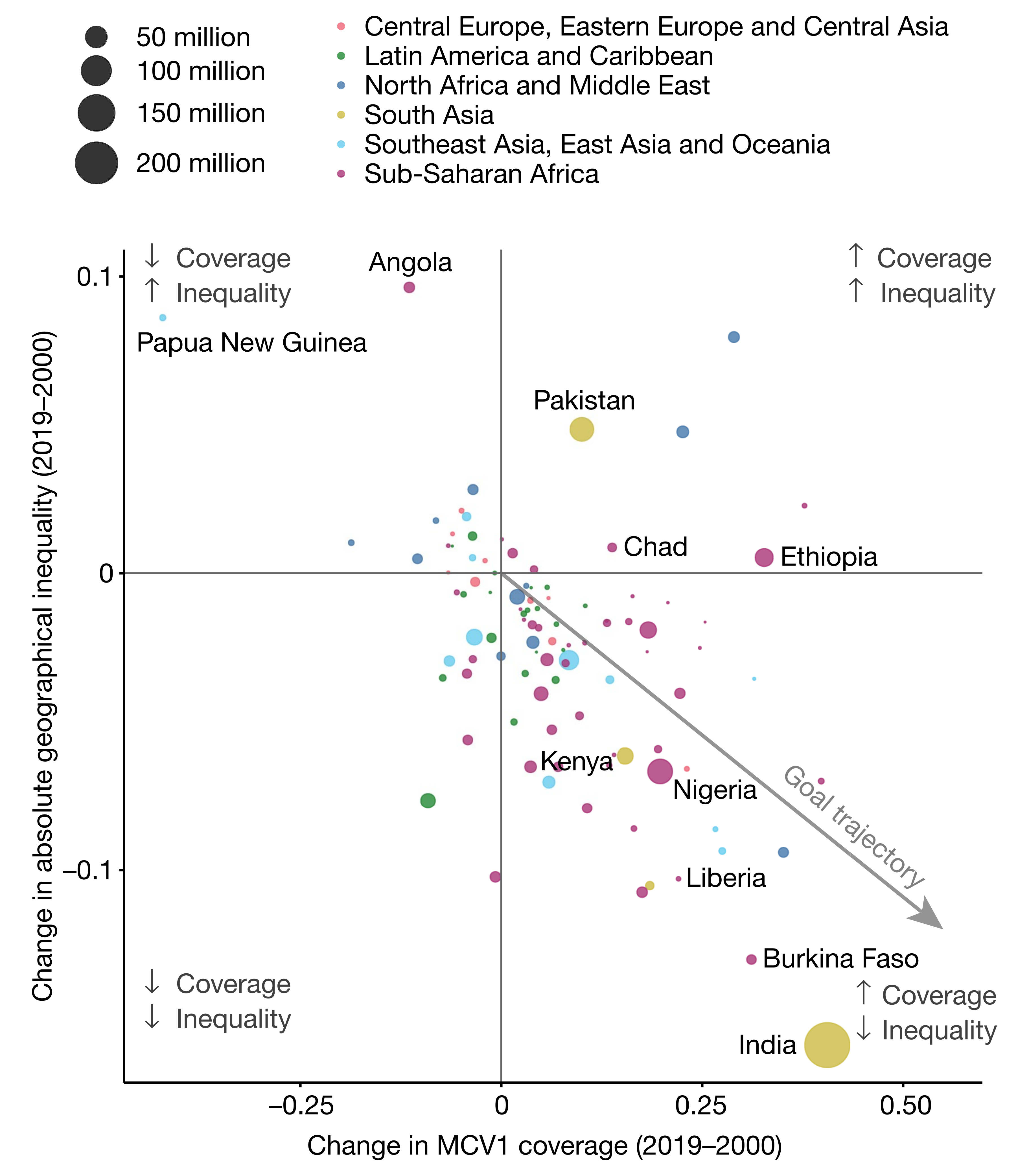
Biểu đồ dưới đây nằm trong bài báo “Mapping routine measles vaccination in low- and middle-income countries”1 được đăng trên tạp chí Nature (IF 2023 = 50.5). Biểu đồ thể hiện tương quan giữa sự thay đổi bất bình đẳng địa lý và sự thay đổi độ phủ vaccine MCV1 của các quốc gia thu nhập thấp và trung bình từ năm 2000 đến năm 2019. Chúng ta sẽ vẽ lại biểu đồ này bằng ggplot2.
6.1.1 Các bước vẽ
Gọi các library cần thiết.
Nhìn vào biểu đồ, chúng ta thấy trục x (nằm ngang) là sự thay đổi độ phủ vaccine, trục y (nằm dọc) là sự thay đổi bất bình đẳng địa lý. Có 10 quốc gia được hiển thị tên là Angola, Papua New Guinea, Pakistan, Chad, Ethiopia, Kenya, Nigeria, Liberia, Burkina Faso, India.
Vậy chúng ta cần một data frame có ít nhất 3 cột: (1) tên quốc gia, (2) sự thay đổi độ phủ vaccine, (3) sự thay đổi bất bình đẳng địa lý.
country <- c("Angola", "Papua New Guinea", "Pakistan", "Chad", "Ethiopia", "Kenya", "Nigeria", "Liberia", "Burkina Faso", "India")
coverage <- c(-0.14, -0.35, 0.12, 0.14, 0.32, 0.07, 0.2, 0.18, 0.3, 0.35)
inequality <- c(0.1, 0.08, 0.05, 0.01, 0.005, -0.06, -0.07, -0.11, -0.13, -0.16)
df_plot <- data.frame(country, coverage, inequality)
df_plotChúng ta sẽ vẽ biểu đồ cho 10 quốc gia này trước. Tưởng tượng nếu vẽ biểu đồ này bằng tay thì mình sẽ vẽ như thế nào? Trước tiên chúng ta vẽ 2 trục tọa độ:
ggplot(df_plot, aes(x = coverage, y = inequality))Sau khi có 2 trục tọa độ, tiến hành chấm các điểm lên theo đúng tọa độ (x, y) của nó. Dạng biểu đồ điểm còn gọi là scatter plot trong ggplot tương ứng với geom_point().
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point()Sau khi có các điểm, chúng ta viết tên quốc gia lên bằng cách dùng geom_text() với label là tên nước (country).
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point() +
geom_text(aes(label = country))Hiện giờ chữ đang đè lên các điểm, chúng ta điều chỉnh vị trí của chữ bằng arguments hjust và vjust của geom_text().
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point() +
geom_text(aes(label = country), hjust = -0.2, vjust = 0.2)Một số tên nước bị cắt mất như Papua New Guinea, chúng ta điều chỉnh lại giới hạn hiển thị của các trục tọa độ bằng xlim(), ylim() để hiển thị lại đầy đủ.
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point() +
geom_text(aes(label = country), hjust = -0.2, vjust = 0.2) +
xlim(c(-0.35, 0.55)) +
ylim(c(-0.17, 0.1))Các điểm trong biểu đồ gốc có màu sắc và kích thước to nhỏ khác nhau, vậy trong data frame cần có thêm 1 cột màu sắc và 1 cột kích thước.
df_plot$size <- c(1.1, 0.6, 3, 1, 2, 1.1, 3, 0.7, 1.1, 8)
df_plot$color <- c("purple", "blue", "yellow", "purple", "purple", "purple", "purple", "purple", "purple", "yellow")
df_plotBây giờ chúng ta có thể thêm màu sắc và kích thước vào geom_point().
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point(aes(size = size, color = color)) +
geom_text(aes(label = country), hjust = -0.2, vjust = 0.2) +
xlim(c(-0.35, 0.55)) +
ylim(c(-0.17, 0.1))Các quốc gia chính đã được vẽ xong, chúng ta cần có các điểm khác trên biểu đồ mà không có tên quốc gia. Có thể tạo ra các điểm ngẫu nhiên như vậy bằng R.
set.seed(123)
np <- 50
rd <- data.frame(
country = rep("", np),
coverage = rnorm(n = np, mean = 0.1, sd = 0.12),
inequality = rnorm(n = np, mean = -0.05, sd = 0.04),
size = rnorm(n = np, mean = 1, sd = 0.4),
color = sample(
c("red", "green", "darkblue", "yellow", "blue", "purple"),
np,
replace = T
)
)
head(rd)set.seed(123): dùng để cố định cách tạo ra các điểm ngẫu nhiên.np <- 50: tạo ra 50 điểm ngẫu nhiên.country = rep("", np): các điểm này không có tên quốc gia.coverage = rnorm(n = np, mean = 0.1, sd = 0.12): sự thay đổi độ phủ vaccine (giá trị trên trục x) của các điểm này là số thực được tạo ra ngẫu nhiên theo phân phối bình thường với trung bình là 0.1 và độ lệch chuẩn là 0.12.inequality = rnorm(n = np, mean = -0.05, sd = 0.04): sự thay đổi bất bình đẳng địa lý (giá trị trên trục y) của các điểm này là số thực được tạo ra ngẫu nhiên theo phân phối bình thường với trung bình là -0.05 và độ lệch chuẩn là 0.04.size = rnorm(n = np, mean = 1, sd = 0.4): kích thước của các điểm này là số thực được tạo ra ngẫu nhiên theo phân phối bình thường với trung bình là 1 và độ lệch chuẩn là 0.4.color = sample(1:6, np, replace = T): màu sắc là số nguyên ngẫu nhiên từ 1 đến 6.
Bây giờ chúng ta ghép data frame rd này với data frame của biểu đồ df_plot. Cột màu sắc được đưa về dạng factor để hiển thị màu. Sau đó dùng code ggplot trên để vẽ.
df_plot <- rbind(df_plot, rd)
df_plot$color <- factor(df_plot$color)
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point(aes(size = size, color = color)) +
geom_text(aes(label = country), hjust = -0.2, vjust = 0.2) +
xlim(c(-0.35, 0.55)) +
ylim(c(-0.17, 0.1))6.1.2 Kết quả
Biểu đồ được tạo ra đã tương đối giống biểu đồ gốc. Chúng ta có thể thêm các đường thẳng x = 0, y = 0, chọn bảng màu, chỉnh lại kích thước các điểm, chọn lại theme cho biểu đồ để nhìn giống biểu đồ gốc hơn.
Code
cols <- c(
"red" = "#fa8495",
"green" = "#4ca258",
"darkblue" = "#6493bb",
"yellow" = "#d7c968",
"blue" = "#7dd8f3",
"purple" = "#bc5c91"
)
ggplot(df_plot, aes(x = coverage, y = inequality)) +
geom_point(aes(size = size, color = color), alpha = 0.8) +
geom_text(aes(label = country), hjust = -0.2, vjust = 0.2) +
geom_vline(xintercept = 0, color = "#999999") +
geom_hline(yintercept = 0, color = "#999999") +
scale_x_continuous(breaks = c(-0.25, 0, 0.25, 0.5),
limits = c(-0.4, 0.55)) +
scale_y_continuous(breaks = c(-0.1, 0, 0.1), limits = c(-0.16, 0.1)) +
scale_size_continuous(
breaks = c(2, 4, 6, 8),
labels = c("50 million", "100 million", "150 million", "200 million"),
range = c(0, 8),
guide = guide_legend(order = 1)
) +
scale_color_manual(
values = cols,
breaks = c("red", "green", "darkblue", "yellow", "blue", "purple"),
labels = c(
"Central Europe, Eastern Europe and Central Asia",
"Latin America and Caribbean",
"North Africa and Middle East",
"South Asia",
"Southeast Asia, East Asia and Oceania",
"Sub-Saharan Africa"
),
guide = guide_legend(order = 2)
) +
labs(x = "Change in MCV1 coverage (2019-2000)",
y = "Change in absolute geographical inequality (2019-2000)",
size = NULL,
color = NULL) +
theme_classic() +
theme(
legend.position = "top",
legend.direction = "vertical",
legend.text = element_text(size = 11),
legend.key.height = unit(0.5, "cm"),
axis.text = element_text(size = 11)
)So sánh với biểu đồ gốc ở đây Figure 6.1.
6.2 Biểu đồ 2
Biểu đồ dưới đây nằm trong bài báo “Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis” được đăng trên tạp chí The Lancet (IF 2023 = 98.4).
6.2.1 Các bước vẽ
Tương tự bài tập trên, chúng ta thấy (1) trục x là các vùng Global Burden of Disease (GBD regions), (2) màu sắc là các GBD super-regions, trục y là số tử vong chia thành 2 loại là (3) associated with resistance và (4) attributable to resistance, mỗi loại số tử vong có (5, 6) cận dưới và (7, 8) cận trên của khoảng tin cậy. Vậy chúng ta phải có data gồm 8 cột như sau.
region <- c(1:21)
sup_region <- c(
"green",
"green",
"green",
"green",
"blue",
"turquoise",
"purple",
"orange",
"purple",
"turquoise",
"red",
"red",
"red",
"orange",
"turquoise",
"purple",
"purple",
"red",
"orange",
"pink",
"purple"
)
assoc <- c(114.8, 89.0, 86, 79.4, 76.8, 74.0, 72.3, 71.6, 70.7, 68.0, 65.1, 63.2, 63.0, 54.8, 53.3, 52.5, 51.0, 50.6, 43.3, 42.0, 28.0)
assoc_up <- c(145.3, 112.6, 109.8, 101.6, 101.2, 105.6, 93.4, 98.0, 92.3, 100.9, 90.5, 85.4, 83.1, 74.9, 74.3, 73.0, 71.5, 70.9, 66.1, 59.5, 39.9)
assoc_lo <- c(90.4, 70.5, 65.9, 61.7, 57.2, 48.8, 55.1, 51.4, 53.2, 43.2, 45.5, 45.4, 47.1, 38.9, 37.7, 37.0, 35.4, 34.7, 27.4, 28.7, 18.8)
attr <- c(27.3, 21.4, 20.7, 19.4, 21.5, 19.9, 18.6, 17.0, 16.5, 16.6, 16.2, 15.9, 15.2, 14.4, 13.8, 11.7, 12.3, 13.0, 10.5, 11.2, 6.5)
attr_up <- c(35.3, 28.1, 27.7, 25.9, 29.8, 28.5, 24.7, 23.2, 23.1, 25.0, 23.2, 21.9, 20.6, 20.0, 19.5, 16.6, 17.5, 18.5, 16.0, 16.3, 9.4)
attr_lo <- c(20.9, 16.3, 14.9, 14.3, 15.1, 13.1, 13.9, 12.1, 11.6, 10.5, 11.0, 11.1, 11.1, 10.0, 9.5, 8.0, 8.3, 8.8, 6.5, 7.5, 4.3)
df_plot <- data.frame(region, sup_region, assoc, assoc_up, assoc_lo, attr, attr_up, attr_lo)
df_plot <- df_plot |>
mutate(region = factor(region),
sup_region = factor(
sup_region,
levels = c("turquoise", "purple", "red", "pink", "blue", "orange", "green")
))Xem data:
head(df_plot)Trước tiên chúng ta vẽ 2 trục tọa độ. Trong trường hợp này, vì trục y vừa là deaths của associated with resistance và attributable to resistance, nên chúng ta có thể chỉ vẽ trục x trước.
ggplot(df_plot, aes(x = region))Chúng ta sẽ vẽ các cột deaths của associated with resistance lên trước. Dạng biểu đồ cột là geom_bar().
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc), stat = "identity")Tô màu cho các cột này.
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region), stat = "identity")Màu chưa hiển thị đúng với màu mình muốn, quy định mã màu và chỉnh màu lại bằng scale_fill_manual()
cols <- c(
"green" = "#a5d474",
"orange" = "#f5b168",
"blue" = "#74accf",
"pink" = "#f7c6dc",
"red" = "#f48073",
"purple" = "#b2b0d5",
"turquoise" = "#82cec2"
)
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region), stat = "identity") +
scale_fill_manual(values = cols)Vẽ khoảng tin cậy bằng geom_errorbar().
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region), stat = "identity") +
geom_errorbar(aes(ymin = assoc_lo, ymax = assoc_up), width = 0.1) +
scale_fill_manual(values = cols)Vậy là đã vẽ được các cột của deaths associated with resistance, giờ chúng ta vẽ deaths attributable to resistance. Một mẹo để 2 cột này chồng lên nhau và hiển thị được một legend cho 2 nhóm này là sử dụng tham số alpha, dùng để quy định độ trong suốt.
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region, alpha = "a"), stat = "identity") +
geom_errorbar(aes(ymin = assoc_lo, ymax = assoc_up), width = 0.1) +
geom_bar(aes(y = attr, alpha = "b"), width = 0.65, stat = "identity") +
geom_errorbar(aes(ymin = attr_lo, ymax = attr_up), width = 0.1) +
scale_fill_manual(values = cols)Việc sử dụng alpha làm cho cột deaths associated with resistance quá trong suốt gần như không thấy gì, nhưng đã hiện legend alpha có 2 màu xám nhạt, xám đậm giống như hình gốc. Tương tự như đã chỉnh màu bằng scale_fill_manual(), chúng ta chỉnh độ trong suốt lại bằng scale_alpha_discrete().
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region, alpha = "a"), stat = "identity") +
geom_errorbar(aes(ymin = assoc_lo, ymax = assoc_up), width = 0.1) +
geom_bar(aes(y = attr, alpha = "b"), width = 0.65, stat = "identity") +
geom_errorbar(aes(ymin = attr_lo, ymax = attr_up), width = 0.1) +
scale_fill_manual(values = cols) +
scale_alpha_discrete(range = c(1, 0.8),
guide = guide_legend(override.aes = list(fill = c("gray70", "gray20"))))6.2.2 Kết quả
Giờ chỉ cần sửa lại tên các vùng, tên cột, tên legend… cho giống với hình gốc.
Code
ggplot(df_plot, aes(x = region)) +
geom_bar(aes(y = assoc, fill = sup_region, alpha = "a"), stat = "identity") +
geom_errorbar(aes(ymin = assoc_lo, ymax = assoc_up), width = 0.1) +
geom_bar(aes(y = attr, alpha = "b"), width = 0.65, stat = "identity") +
geom_errorbar(aes(ymin = attr_lo, ymax = attr_up), width = 0.1) +
scale_x_discrete(
expand = c(0, 0),
labels = c(
"Western sub-Saharan Africa",
"Eastern sub-Saharan Africa",
"Central sub-Saharan Africa",
"Southern sub-Saharan Africa",
"South Asia",
"Eastern Europe",
"Southern Latin America",
"Oceania",
"High-income North America",
"Central Europe",
"Caribbean",
"Andean Latin America",
"Tropical Latin America",
"Southeast Asia",
"Central Asia",
"Western Europe",
"High-income North America",
"Central Latin America",
"East Asia",
"North Africa and Middle East",
"Australasia"
)
) +
scale_y_continuous(
limits = c(0, 165),
breaks = c(0, 50, 100, 150),
expand = c(0, 0)
) +
scale_fill_manual(
values = cols,
labels = c(
"Central Europe, eastern Europe, and central Asia",
"High income",
"Latin America and Caribbean",
"North Africa and Middle East",
"South Asia",
"Southern Asia, east Asia, and Oceania",
"Sub-Saharan Africa"
),
guide = guide_legend(order = 1)
) +
scale_alpha_discrete(
range = c(1, 0.8),
guide = guide_legend(override.aes = list(fill = c("gray70", "gray20")), order = 2),
labels = c("Associated with resistance", "Attributable to resistance")
) +
labs(x = "GBD region",
y = "Deaths (rate per 100000 population)",
fill = "GBD super-region",
alpha = "Resistance") +
theme_classic() +
theme(
axis.text = element_text(size = 11),
axis.text.x = element_text(angle = 45, hjust = 1),
legend.position = c(0.57, 0.86),
legend.box = "horizontal",
legend.key.height = unit(0.5, "cm"),
legend.text = element_text(size = 10),
legend.title = element_text(face = "bold"),
plot.margin = margin(10, 10, 10, 60)
)So sánh với biểu đồ gốc ở đây Figure 6.2.
6.3 Bài tập
6.3.1 Bài 1
Dùng data thực hành simulated_covid.rds để vẽ biểu đồ cột thể hiện số ca nhiễm ở từng quận.
df <- readRDS("data/simulated_covid.rds")Gợi ý: Đếm số ca nhiễm ở từng quận bằng lệnh count().
df_plot <- df |>
count(district)6.3.2 Bài 2
Vẽ biểu đồ sau bằng data thực hành ex-nature.xlsx.
Đọc và xử lý data.
library(readxl)
library(stringr)
df <- read_excel("data/ex-nature.xlsx")
# Bars
df_bar <- data.frame(df[-1, 1:6])
colnames(df_bar) <- as.character(df[1, 1:6])
df_bar <- df_bar |>
mutate_at(vars(1:6), as.numeric) |>
pivot_longer(cols = starts_with("on"), values_to = "value") |>
mutate(name = str_to_sentence(name)) |>
as.data.frame()
# Lines
df_line <- data.frame(df[-1, c(1, 7:9)])
colnames(df_line) <- as.character(df[1, c(1, 7:9)])
df_line <- df_line |>
mutate_at(vars(1:4), as.numeric)Code
ggplot() +
geom_bar(
data = df_bar,
mapping = aes(
x = week,
y = value,
fill = factor(
name,
levels = c(
'Onset >15 days before testing',
'Onset 8-15 days before testing',
'Onset 5-7 days before testing',
'Onset 2-4 days before testing',
'Onset 0-1 days before testing'
)
)
),
stat = "identity"
) +
scale_fill_manual(
values = c(
"Onset 0-1 days before testing" = "#fcd0a1",
"Onset 2-4 days before testing" = "#fead6c",
"Onset 5-7 days before testing" = "#fd8d3a",
"Onset 8-15 days before testing" = "#f36912",
"Onset >15 days before testing" = "#da4800"
)
) +
geom_hline(yintercept = 100.3, linewidth = 1) +
scale_x_continuous(breaks = seq(20, 26, by = 1)) +
geom_line(
data = df_line,
mapping = aes(x = week, y = median * 100 / 14),
linetype = "dashed",
color = "#810004"
) +
geom_point(data = df_line,
aes(x = week, y = median * 100 / 14),
color = "#810004") +
geom_errorbar(
data = df_line,
aes(
x = week,
ymin = `95% CI lower` * 100 / 14,
ymax = `95% CI upper` * 100 / 14
),
width = 0,
color = "#810004"
) +
scale_y_continuous(
name = "Laboratory-confirmed \n cases showing symptoms (%)",
breaks = seq(0, 100, by = 20),
expand = c(0, 0),
sec.axis = sec_axis(
transform = ~ . / (100 / 14),
name = "Delay from onset to testing",
breaks = seq(0, 14, by = 2)
)
) +
labs(x = "Week of testing", fill = NULL) +
theme_classic() +
theme(
axis.title.y.right = element_text(angle = 90, color = "#78070b"),
legend.position = "bottom",
legend.direction = "vertical",
legend.key.height = unit(0.1, "cm"),
legend.key.width = unit(0.6, "cm"),
legend.key.spacing.y = unit(0.1, "cm"),
legend.text = element_text(size = 11)
)So sánh với biểu đồ gốc ở đây Figure 6.3.