library(tidyverse)
library(sf)
library(plotly)
library(leaflet)7 Trực quan hóa 2
Hiểu cách sử dụng các lệnh
geom_*()của ggplotSử dụng plotly để tạo biểu đồ tương tác
Học cách vẽ bản đồ bằng leaflet
7.1 ggplot
Chúng ta sẽ sử dụng bộ data Covid từ phần phân tích cho các ví dụ trong bài học này
linelist <- read_rds("data/simulated_covid.rds") %>% mutate_if(is.Date, ymd) 7.1.1 Ôn tập ggplot
Như đã học ở phần trước, quá trình vẽ ggplot được thực hiện tương tự như quá trình vẽ đồ thị thủ công, bao gồm việc vẽ từng lớp đồ thị và các lớp sẽ được nối bằng dấu +.
Quá trình vẽ như sau
Bắt đầu với lớp nền bằng lệnh
ggplot()- nơi người dùng thường quy định dữ liệu được vẽ và các biến nằm trên trục x và yThêm các lớp
geom- mỗi lớp geom sẽ thêm 1 loại biểu đồ (VD: biểu đồ cột, biểu đồ đường, biểu đồ phân tán, histogram). Các hàm này đều bắt đầu bằnggeom_*Thêm các yếu tố thiết kế vào đồ thị, chẳng hạn như nhãn trục, tiêu đề, phông chữ, kích thước, phối màu, chú giải hoặc xoay trục
VD: Vẽ số ca theo thời gian từ dữ liệu covid
# ------ Tính số ca theo ngày phát triệu chứng ------
case_data <- linelist %>%
group_by(date_onset) %>% # nhóm theo ngày phát triệu chứng
summarize(
no_cases = n() # đếm số ca theo nhóm
)
# ------- Vẽ biểu đồ bằng ggplot() ------
ggplot(
data = case_data, # sử dụng case_data để plot
# quy định biến sử dụng làm trục x và y
# lấy ngày phát triệu chứng làm trục x, số ca làm trục y
aes(x = date_onset, y = no_cases)
) +
geom_col( # vẽ biểu đồ cột
# thêm các tuỳ chọn khác, ở vd này quy định màu là "cornflowerblue"
color = "cornflowerblue"
)+
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)+
theme_minimal() # chỉnh thêm màu sắc, font size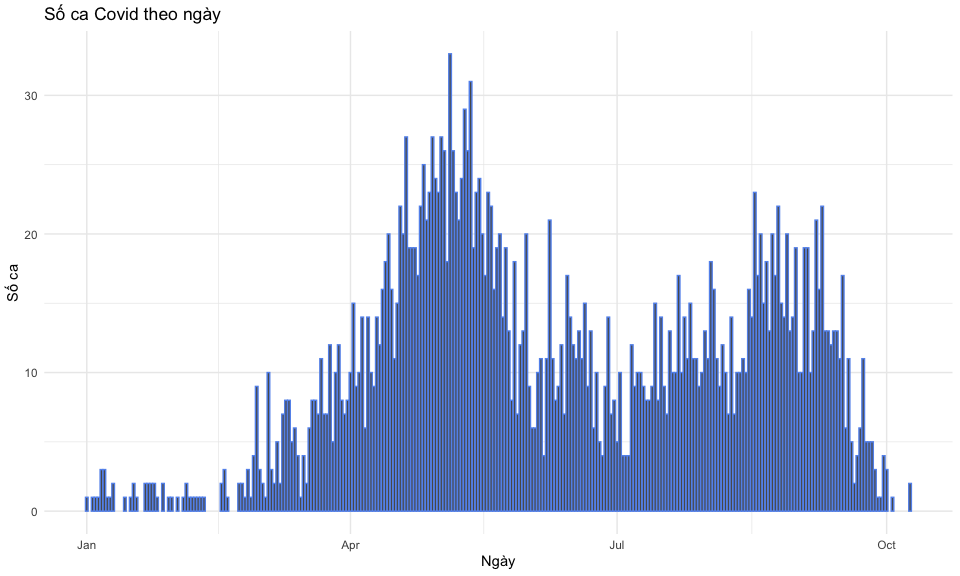
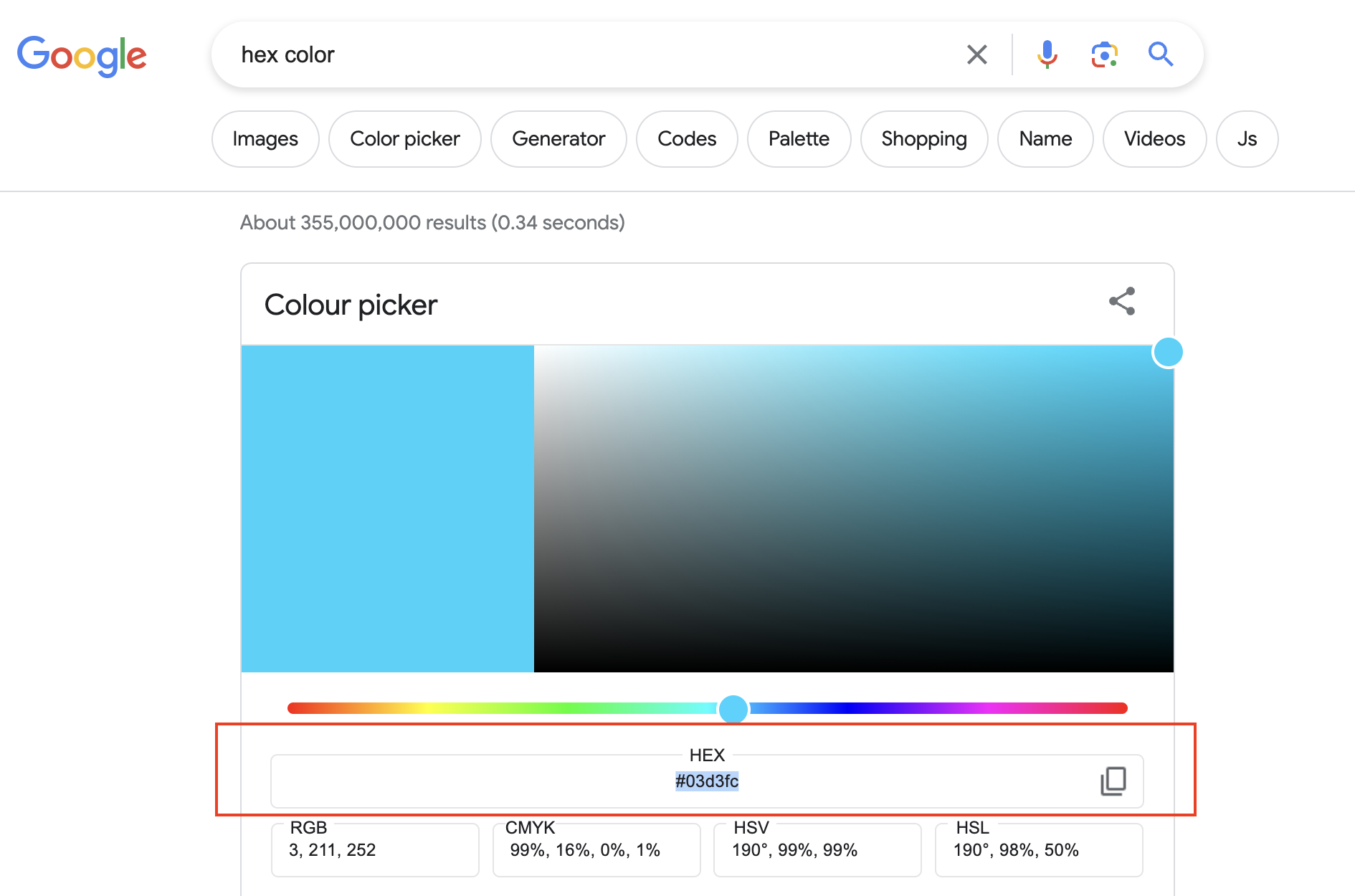
Ngoài các màu cơ bản, ta có thể quy định màu trong R dưới dạng hex color
VD
ggplot(data = my_data)+
geom_point(
aes(x = col1, y = col2),
color = "#03d3fc")Để có được hex color, ta có thể google “hex color”, chọn màu phù hợp và copy HEX
7.1.2 Một số geom_ thông dụng
Lệnh geom_ |
Công dụng | Các tuỳ chỉnh |
|---|---|---|
geom_histogram() |
Tạo biểu đồ cột với x là biến liên tục động được chia thành các khoảng (bin) còn y là số quan sát trong mỗi khoảng |
Dữ liệu cần cung cấp: Giá trị cho cột
|
geom_col() |
Tạo biểu đồ cột từ x và y | Dữ liệu cần cung cấp: Giá trị cho cột x và y |
geom_bar() |
Tương tự như geom_col() nhưng y sẽ tự động được tính theo tham số stat |
Dữ liệu cần cung cấp: Giá trị cho cột
|
geom_boxplot() |
Tạo boxplot từ x và y (nếu 1 trục là biến liên trục, trục còn lại phải thuộc biến phân loại) | Dữ liệu cần cung cấp: Giá trị cho cột x và y |
geom_point() |
Tạo biểu đồ phân tán từ x và y | Dữ liệu cần cung cấp: Giá trị cho cột x và y |
geom_line() |
Tạo biểu đồ đường từ x và y | Dữ liệu cần cung cấp: Giá trị cho cột x và y |
geom_smooth() |
Tạo biểu đồ đường hồi quy cùng khoảng tin cậy (confidence interval) từ x và y | Dữ liệu cần cung cấp: Giá trị cho cột
|
- Histograms -
geom_histogram()
Code geom_histogram()
ggplot(
data = case_data, aes(x = date_onset) ) +
geom_histogram(
fill = "cornflowerblue"
) +
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.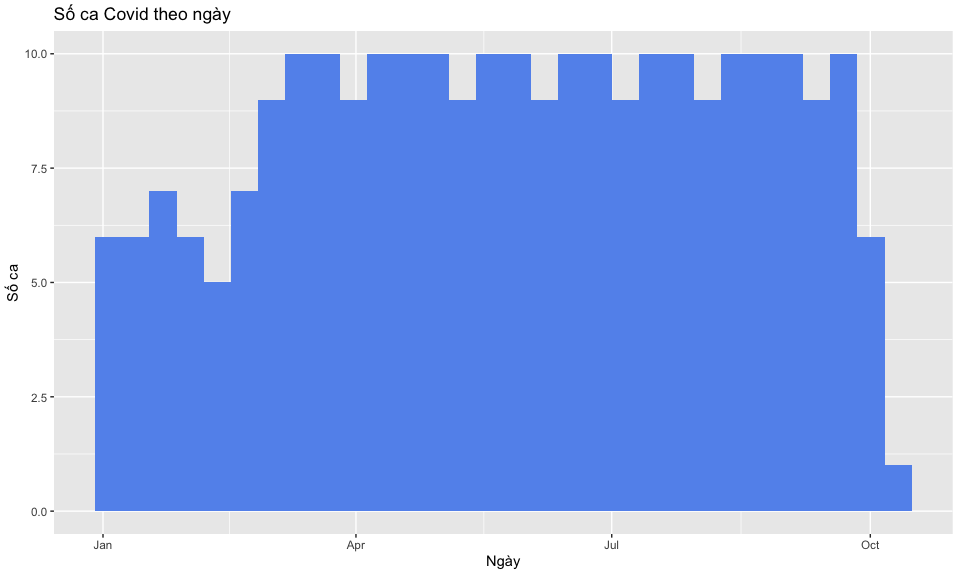
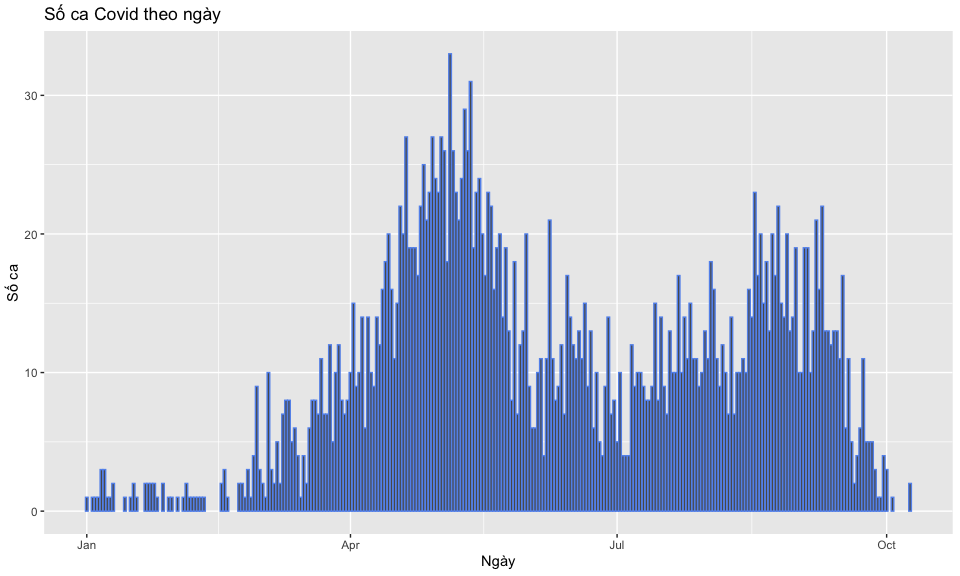
- Biểu đồ cột -
geom_bar()hoặcgeom_col()
Code geom_col()
ggplot(
data = case_data,
aes(x = date_onset, y = no_cases)
) +
geom_col(
color = "cornflowerblue"
)+
labs(
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)
- Box plots -
geom_boxplot()
Code geom_boxplot()
# --- Tính số ca mỗi ngày trong từng đợt dịch ----
linelist %>%
group_by(outbreak, date_onset) %>%
summarize(
no_cases = n()
) %>%
# --- Vẽ boxplot so sánh số ca mỗi ngày trong 2 đợt dịch ----
ggplot(
aes(x = outbreak, y = no_cases) ) +
geom_boxplot() +
labs(
x = "Đợt dịch",
y = "Số ca mỗi ngày"
)`summarise()` has grouped output by 'outbreak'. You can override using the
`.groups` argument.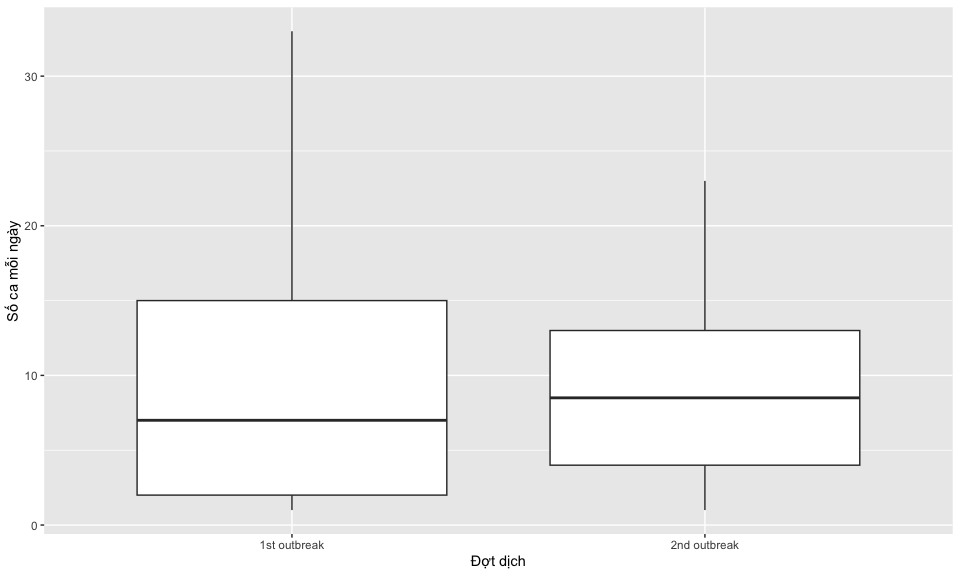
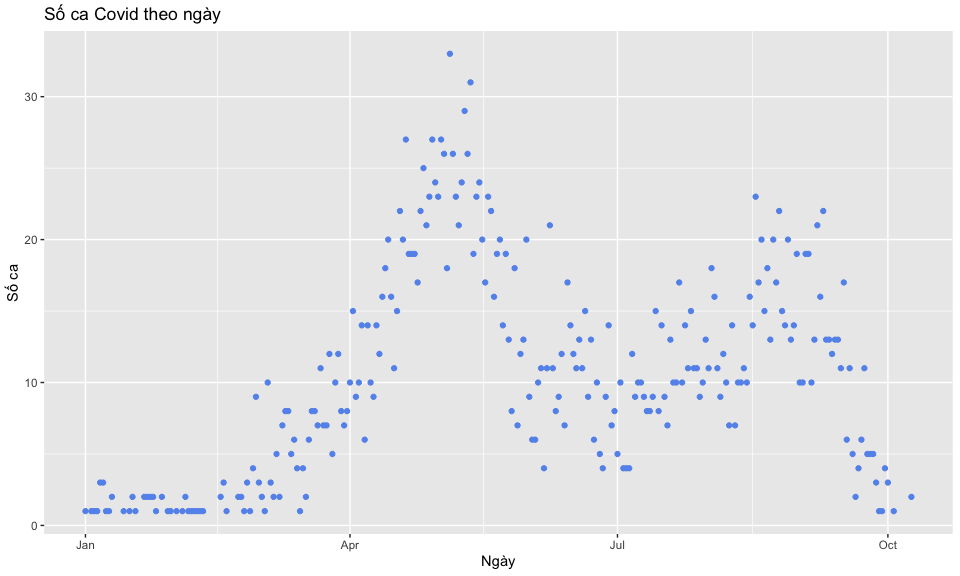
- Điểm (vd: biểu đồ phân tán) -
geom_point()
Code geom_point()
ggplot(
data = case_data, aes(x = date_onset, y = no_cases) ) +
geom_point(
color = "cornflowerblue"
) +
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)
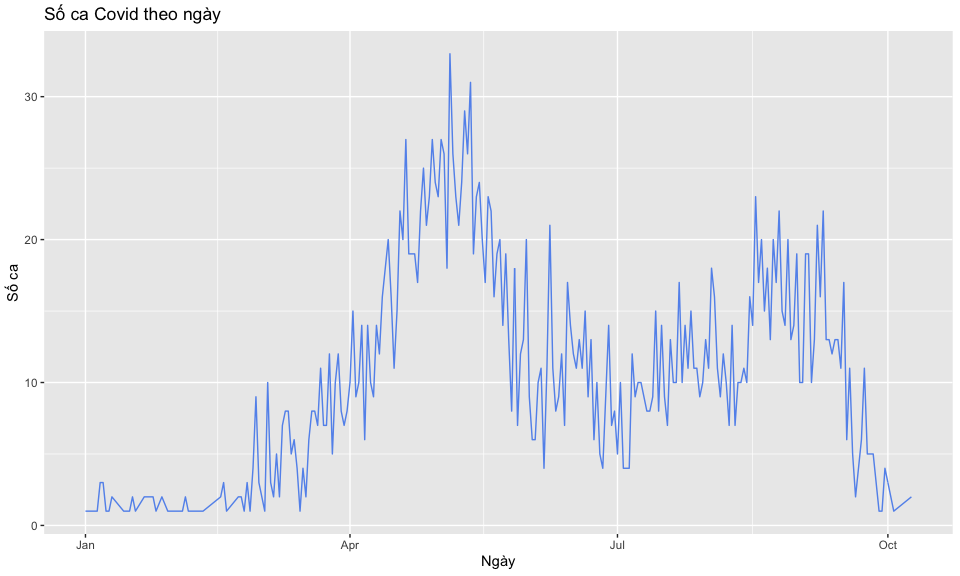
- Biểu đồ đường -
geom_line()
Code geom_line()
ggplot(
data = case_data, aes(x = date_onset, y = no_cases) ) +
geom_line(
color = "cornflowerblue"
) +
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)
- Đường xu hướng -
geom_smooth()
Code geom_smooth()
ggplot(
data = case_data, aes(x = date_onset, y = no_cases) ) +
geom_smooth(
color = "cornflowerblue"
) +
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)`geom_smooth()` using method = 'loess' and formula = 'y ~ x'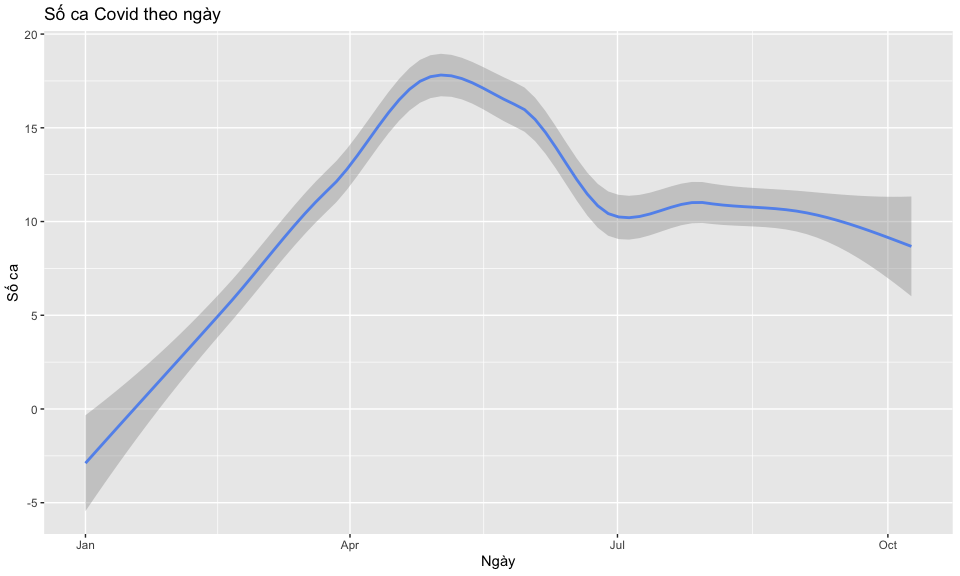
Code geom_area()
ggplot(
data = case_data, aes(x = date_onset, y = no_cases) ) +
geom_area(
fill = "cornflowerblue", color = "black"
) +
labs(
# thêm tên bảng và tên các trục
title = "Số ca Covid theo ngày",
x = "Ngày",
y = "Số ca"
)
stat của hàm geom
"identity"lấy dữ liệu thô"count"đếm số quan sát (số hàng) trong từng phân nhóm"sum"tính tổng của các hàng trong từng phân nhóm
VD: vẽ biểu đồ số ca trực tiếp từ data linelist thay vì case_data bằng các quy định stat = count
ggplot(
data = linelist,
aes(x = date_onset) # không cần quy định y ở đây
) +
geom_bar(
# ggplot sẽ tự tính y bằng các đếm số hàng nhóm theo mỗi giá trị của x (đếm số ca theo date_onset)
stat = "count",
color = "cornflowerblue"
) +
labs(title = "Số ca Covid theo ngày",
x = "Ngày", y = "Số ca"
) 
7.1.3 Điều chỉnh các đặc điểm của biểu đồ
Một số đặc điểm quan trọng cho các lớp geom_
| Tên đặc điểm | Công dụng |
|---|---|
fill |
quy định fill color cho đồ thị |
color |
quy định border color cho đồ thị |
size |
quy định kích cỡ của đường/điểm cho đồ thị |
alpha |
quy định độ trong suốt của màu (từ 0-1, càng gần 0 màu càng trong suốt) |
linetype |
quy định loại đường (thẳng, đứt, …) cho các đồ thị đường |
shape |
quy định loại điểm cho các đồ thị có điểm |
Ta có thể quy định giá trị cho các đặc điểm của biểu đồ như một tham số của lệnh geom_ (VD: geom_bar(color = "cornflowerblue") trong các ví dụ trên)
Tuy nhiên, nếu muốn thay đổi các đặc điểm dựa theo biến từ dữ liệu, ta cần quy định mối liên hệ giữa biến và đặc điểm trong lệnh aes()
aes()
Lệnh aes được sử dụng để quy định mối quan hệ giữa các biến và các đặc điểm của biểu đồ
VD: quy định màu của cột theo đợt dịch
linelist %>%
mutate(
# ----- Đổi label cho từng đợt dịch ------
outbreak = factor(outbreak,
levels = c("1st outbreak", "2nd outbreak"),
labels = c("Đợt dịch 1", "Đợt dịch 2"))
) %>%
ggplot(
aes(x = date_onset)
) +
geom_bar(
aes(fill = outbreak) # quy định mỗi đợt dịch có màu khác nhau
) +
labs(title = "Số ca Covid theo ngày",
x = "Ngày", y = "Số ca",
# optional: đổi tên ô chú thích
fill = "Đợt dịch"
) 
Để tự quy định màu cho biểu đồ, ta có thể sử dụng các lệnh scale_color_*() khi muốn quy định màu viền, hoặc scale_fill_*() quy định màu fill
| Function | Công dụng | Tham số |
|---|---|---|
|
Quy định màu cho từng giá trị biến phân loại |
|
|
Quy định màu cho biến liên tục |
|
|
Tạo thang màu bằng cách chia giá trị biến liên tục thành các nhóm (bins). |
|
VD: quy định màu và label cho từng đợt dịch bằng lệnh scale_fill_manual()
linelist %>%
ggplot(aes(x = date_onset)) +
geom_bar(aes(fill = outbreak) ) +
# ------ Quy định màu cho từng đợt dịch ------
scale_fill_manual(
breaks = c("1st outbreak", "2nd outbreak"),
values = c("#91deed", "#67b6e0"),
labels = c("Đợt dịch 1", "Đợt dịch 2")
) +
labs(title = "Số ca Covid theo ngày", x = "Ngày", y = "Số ca", fill = "Đợt dịch") 
7.1.4 Facet
Facets, hay “chia nhỏ biểu đồ”, được sử dụng để chia một biểu đồ thành nhiều phần nhỏ, với mỗi phần (“facet”) đại diện cho một nhóm của dữ liệu.
Trong ggplot, có 2 loại facet chính
facet_wrap()hiện thị các biểu đồ khác nhau cho từng nhóm của một biến số. (VD: thể hiện các đường cong dịch bệnh khác nhau cho từng khu vực).facet_grid()áp dụng khi muốn đưa một biến thứ hai vào sắp xếp các biểu đồ con. Ở đây mỗi ô thể hiện sự giao nhau của các giá trị giữa hai cột.
| facet_wrap | facet_grid |
|---|---|
| biểu đồ số ca bệnh sốt rét chia theo tỉnh (tên tỉnh ở phía trên mỗi biểu đồ) | biểu đồ số ca bệnh sốt rét chia theo tỉnh và nhóm tuổi |
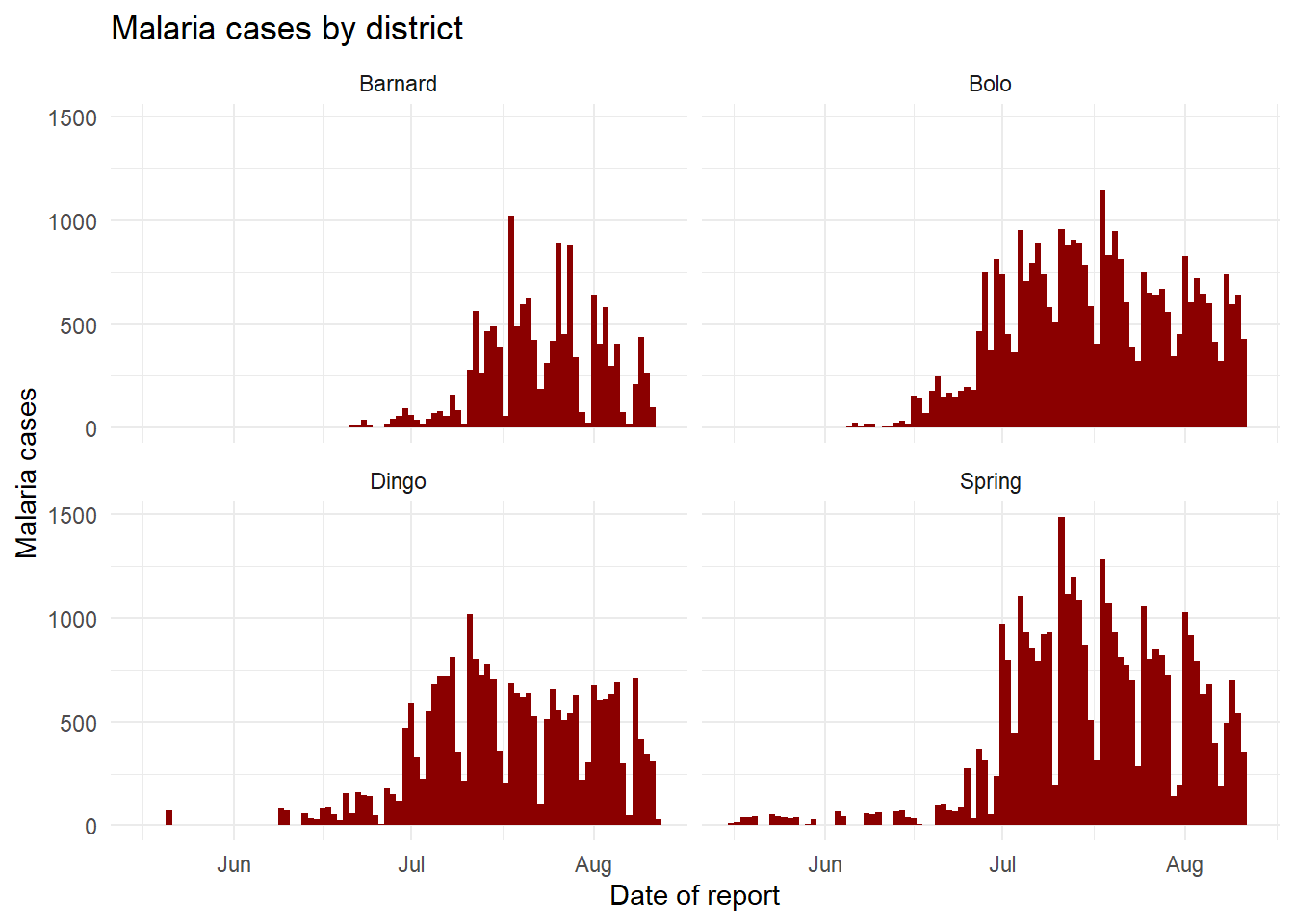 |
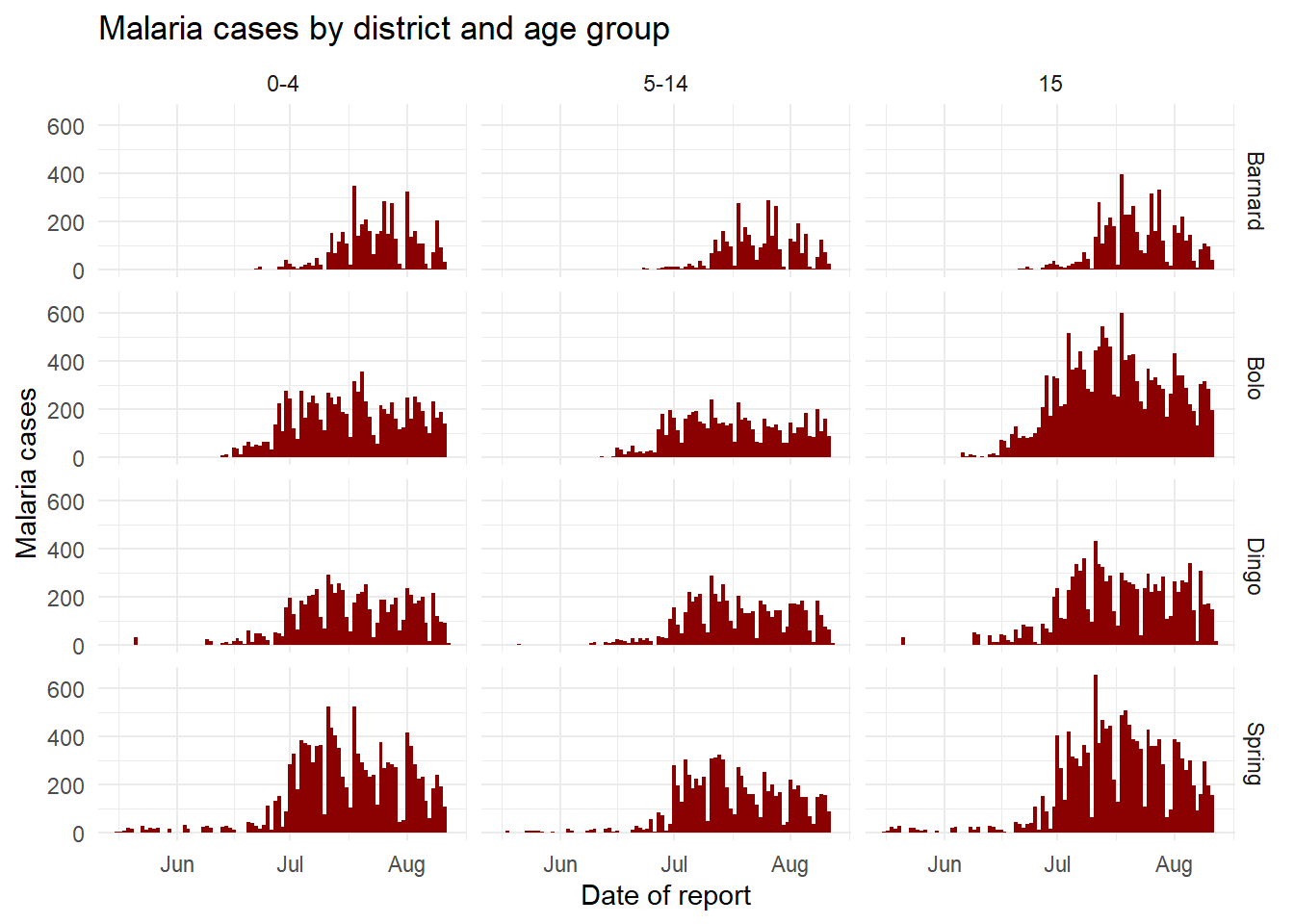 |
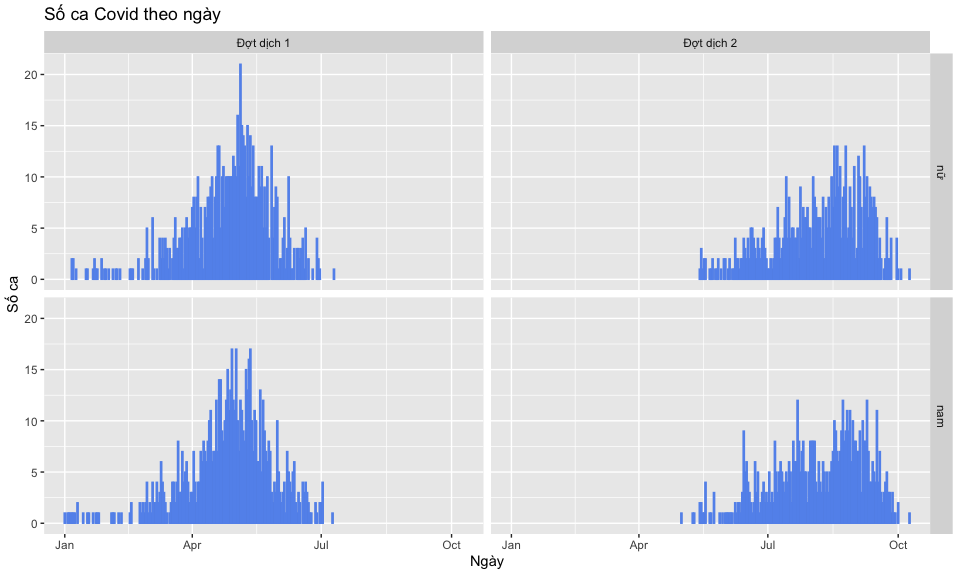
VD: dùng facet_grid để vẽ biểu đồ theo giới tính và đợt dịch
linelist %>%
mutate(
# optional: đổi label cho biến đợt dịch và giới tính
outbreak = factor(outbreak,
levels = c("1st outbreak", "2nd outbreak"),
labels = c("Đợt dịch 1", "Đợt dịch 2")),
sex = factor(sex,
levels = c("f", "m"),
labels = c("nữ", "nam"))
) %>%
ggplot(
aes(x = date_onset)
) +
geom_bar(
color = "cornflowerblue"
) +
labs(title = "Số ca Covid theo ngày",
x = "Ngày", y = "Số ca"
) +
facet_grid(
cols = vars(outbreak),
rows = vars(sex)
)
7.1.5 Lưu biểu đồ
Để lưu biểu đồ, ta sử dụng lệnh ggsave() với các tham số sau
pathđường dẫn đến nơi lưu bản đồ
filenametên file được lưuplotbiểu đồ được lưu (tự động lưu biểu đồ được vẽ gần nhất)width,heightkích cỡ của ảnh được lưudpiđộ phân giải của ảnh được lưu
VD: lưu biểu đồ
ggsave(
path = file.path(getwd(), "images"), # lưu tại folder images
filename = "latest_plot.png", # lưu với tên latest_plot.png
width = 3000, height = 2100, # chỉnh size ảnh
units = "px", # quy định kích cỡ size theo pixel
create.dir = TRUE, # tạo folder nếu folder không tồn tại
dpi = 400)7.2 Plotly
Package plotly cung cấp lệnh ggplotly giúp người dùng nhanh chóng biến biểu đồ của ggplot thành biểu đồ tương tác (interactive plot).
VD: dùng ggplotly để biến biểu đồ số ca thành dạng tương tác
# Vẽ biểu đồ bằng ggplot
case_plot <- linelist %>%
mutate(
# ----- Đổi label cho từng đợt dịch ------
outbreak = factor(outbreak,
levels = c("1st outbreak", "2nd outbreak"),
labels = c("Đợt dịch 1", "Đợt dịch 2"))
) %>%
group_by(outbreak, date_onset) %>%
summarize(no_cases = n()) %>%
ggplot(aes(x = date_onset, y = no_cases)) +
geom_col( aes(fill = outbreak) ) +
scale_x_date() +
labs(title = "Số ca Covid theo ngày", x = "Ngày", y = "Số ca", fill = "Đợt dịch") `summarise()` has grouped output by 'outbreak'. You can override using the
`.groups` argument.# Dùng lệnh ggplotly để biến thành biểu đồ tương tác
ggplotly(case_plot)Khi knit file .Rmd sang các định dạng file khác, tính tương tác của đồ thị chỉ còn khi người dùng knit thành file .html
Để thay đổi giá trị hiển trị trên tooltip, ta có thể tạo một đặc điểm qua lệnh aes() và quy định giá trị cho tham số tooltip khi sử dụng plotly
example_plot <- linelist %>%
mutate(
# ----- Đổi label cho từng đợt dịch ------
outbreak = factor(outbreak,
levels = c("1st outbreak", "2nd outbreak"),
labels = c("Đợt dịch 1", "Đợt dịch 2"))
) %>%
group_by(outbreak, date_onset) %>%
summarize(no_cases = n()) %>%
ggplot(
aes(x = date_onset, y = no_cases,
# ------- Tạo đặc điểm mới cho tooltip, đặt tên là text ------
text = paste0(
"Ngày: ", date_onset,
"\nSố ca: ",no_cases,
"\nĐợt dịch: ", outbreak
))
) +
geom_col( aes(fill = outbreak) ) +
scale_x_date() +
labs(title = "Số ca Covid theo ngày", x = "Ngày", y = "Số ca", fill = "Đợt dịch") `summarise()` has grouped output by 'outbreak'. You can override using the
`.groups` argument.ggplotly(
example_plot,
# quy định lấy đặc điểm text làm tooltip
tooltip = "text"
)7.3 Vẽ bản đồ
7.3.1 Dữ liệu bản đồ
Trước tiên, ta cần dữ liệu để vẽ biểu đồ trong R. Trong khoá học này ta sẽ sử dụng dữ liệu được cấp bởi GADM.
Để download dữ liệu biểu đồ Việt Nam, thực hiện các bước sau:
Vào trang https://gadm.org/download_country.html
Chọn
Vietnamtrong danh sách CountryBấm
GeopackageFile được tải về có tên là
gadm41_VNM.gpkg
Các nguồn bản đồ mở thường sẽ không có các phần lãnh thổ tranh chấp (VD: Hoàng Sa, Trường Sa)
Các thay đổi về bản đồ hành chính có thể không được cập nhật kịp thời (VD: các quận được sáp nhập thành TP. Thủ Đức)
Dữ liệu bản đồ tải về sẽ có 3 cấp bậc
Cấp tỉnh/ thành phố
Cấp quận/ huyện
Cấp phường/ xã
Để đọc dữ liệu bản đồ, dùng lệnh st_read của package sf
Để vẽ dữ liệu đọc được, dùng lệnh geom_sf của ggplot
# Đọc dữ liệu bản đồ
map_path <- "data"
vn_tinh <- st_read(dsn = file.path(map_path, "gadm41_VNM.gpkg"), layer = "ADM_ADM_1")
vn_qh <- st_read(dsn = file.path(map_path, "gadm41_VNM.gpkg"), layer = "ADM_ADM_2")
vn_px <- st_read(dsn = file.path(map_path, "gadm41_VNM.gpkg"), layer = "ADM_ADM_3")# Vẽ dữ liệu đọc được
ggplot(vn_tinh) + geom_sf()
ggplot(vn_qh) + geom_sf()
ggplot(vn_px) + geom_sf()
Nếu gặp lỗi như sau khi chạy geom_sf()
In CPL_crs_from_input(x) :
GDAL Error 1: PROJ: proj_create_from_database: Cannot find proj.dbChạy dòng code sau để giải quyết lỗi (@edzer)
remove.packages("sf")
install.packages('sf', repos = c('https://r-spatial.r-universe.dev'))
library(sf)7.3.2 Điều chỉnh dữ liệu bản đồ
Khi thử nhìn dữ liệu cho bản đồ, ta có thể thấy dữ liệu đọc được cũng là một bảng dữ liệu và nhiều lệnh điều chỉnh bảng dữ liệu cũng có thể áp dụng với bảng này
Một số cột đáng lưu ý bao gồm
Các cột có format
NAME_`cấp bậc hành chính`chứa tên tiếng ViệtCác cột có format
VARNAME_`cấp bậc hành chính`chứa tên tiếng AnhCột
geomsẽ chứa dữ liệu để vẽ nên bản đồ
head(vn_qh)Để lựa chọn các khu vực được vẽ trên bản đồ, ta có thể sử dụng lệnh filter() tương tự như các bảng dữ liệu khác
VD: chỉ vẽ bản đồ các quận của phố Hồ Chí Minh
hcm_map <- vn_qh %>% filter( NAME_1 == "Hồ Chí Minh" )
hcm_map %>% ggplot() + geom_sf()
Để nối một số khu vực được vẽ trên bản đồ, ta có thể sử dụng lệnh st_join() của gói sf
VD: gộp quận 2, quận 9 và quận Thủ Đức thành Thành phố Thủ Đức
# quy định các quận được gộp
thuduc_subset <- (hcm_map$VARNAME_2 == "District 2" | hcm_map$VARNAME_2 == "District 9" |
hcm_map$VARNAME_2 == "Thu Duc" )
# gộp các quận 2, 9, Thủ Đức và lưu giá trị vào cột geom của Thủ Đức trong bảng
hcm_map[hcm_map$VARNAME_2 == "Thu Duc", "geom"] <- st_union( subset(hcm_map, thuduc_subset)$geom )
# xoá dữ liệu cho Q2 và Q9 sau khi gộp
hcm_map <- hcm_map %>% filter(!VARNAME_2 == "District 2" & !VARNAME_2 == "District 9")
# vẽ biểu đồ sau khi gộp
hcm_map %>% ggplot() + geom_sf()
Để thêm giá trị từ một bảng khác vào bảng dữ liệu biểu đồ, ta có thể sử dụng left_join() tương tự các bài học trước
VD: vẽ bản đồ cùng số ca
# ------- Tính tổng số ca -------
case_by_district <- linelist %>%
group_by(district) %>%
summarize(
no_cases = n()
)
# -------- Nối bảng số ca và bảng dữ liệu bản đồ -----
hcm_map <- hcm_map %>%
mutate(
# Trước khi join, phải điều chỉnh cột dùng để join để các giá trị tương ứng giống nhau
VARNAME_2 = str_replace(VARNAME_2, "District", "Quan")
) %>%
left_join(
case_by_district,
by = join_by(VARNAME_2 == district)
)
# --------- Vẽ biểu đồ cùng dữ liệu số ca bằng ggplot ------
hcm_map %>%
ggplot() +
geom_sf(
# quy định màu theo số ca
aes(fill = no_cases)
) +
labs(fill = "Số ca")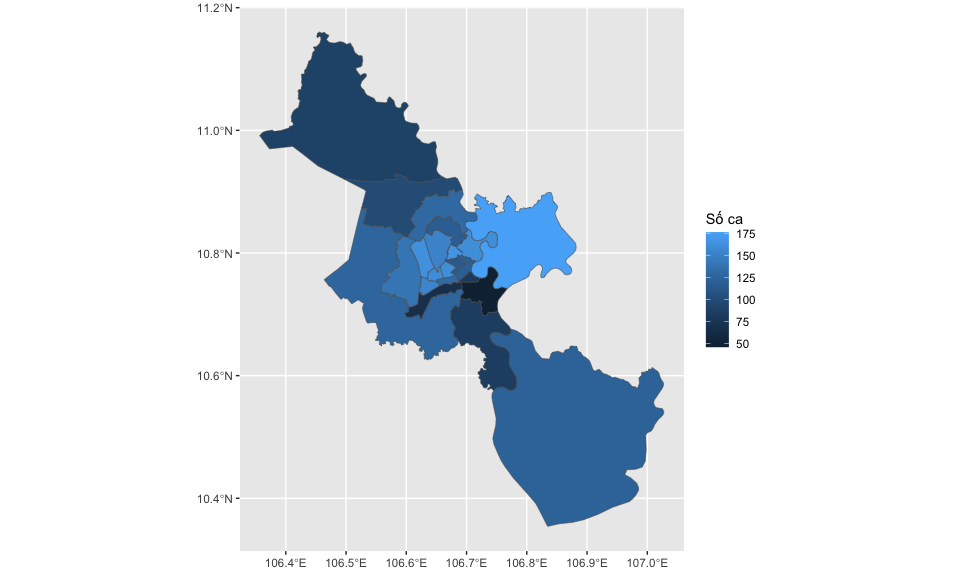
7.3.3 Leaflet
Gói leaflet được dùng để vẽ bản đồ tương tác.
Chúng ta sử dụng leaflet thay vì ggplot + plotly vì bản đồ vẽ bởi leaflet thường đẹp hơn
Quá trình vẽ biểu đồ bằng leaflet cũng tương tự như ggplot, bao gồm các bước sau:
Gọi
leaflet()và cung cấp dữ liệu cần vẽThêm lớp biểu đồ qua lệnh
addPolygon()Thêm ô chú thích bằng lệnh
addLegend()nếu cần thiết
| Tên lệnh | Các tham số thông dụng |
|---|---|
addPolygon() |
|
addLegend() |
|
# ----- quy định bảng màu ----
pal <- colorBin(
# bảng màu cho giá trị từ nhỏ đến lớn
palette = c("#cadefa","#73aeff", "#134c9c"),
domain = hcm_map$no_cases, # cung cấp khoảng giá trị
na.color = "grey" # quy định màu cho giá trị trống
)
# ----- vẽ biểu đồ ------
leaflet(hcm_map) %>%
# vẽ biểu đồ
addPolygons(
color="black", # quy định màu viền
# quy định fillColor dựa theo số ca, và dùng màu từ bảng màu pal
fillColor = ~pal(no_cases),
weight = 1, # độ dày của viền
# chỉnh tooltip
label = ~paste0(NAME_2, ". \nSố ca: ", no_cases),
fillOpacity=0.8
) %>%
# thêm ô chú thích
addLegend(pal = pal, values = ~no_cases, title = "Số ca", position = "bottomright", opacity=1)| Function | Công dụng |
|---|---|
colorNumeric() |
tạo palette màu liên tục từ biến liên tục |
colorBin() |
tạo palette màu phân loại từ biến liên tục |
colorFactor() |
tạo palette màu phân loại từ biến phân loại |
7.4 Bài tập
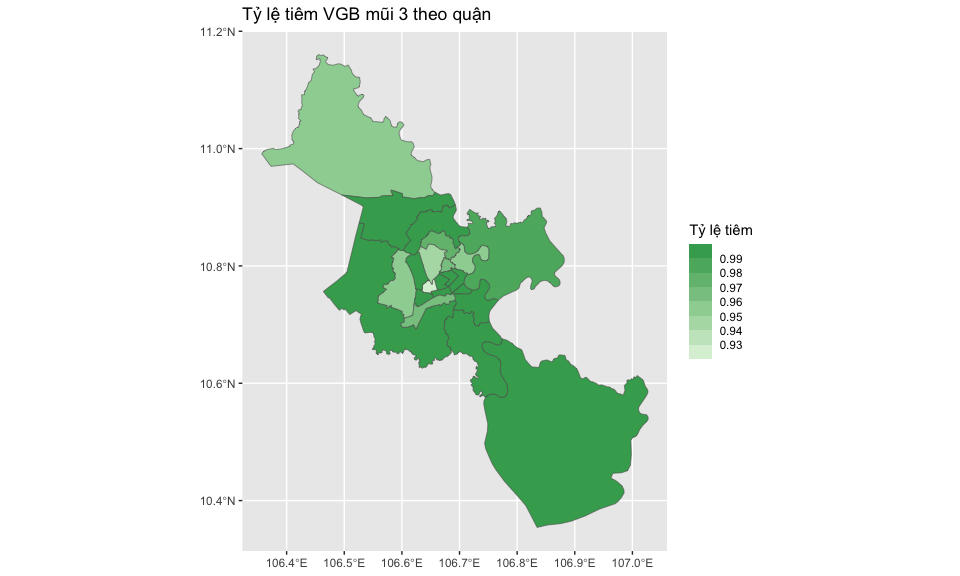
Đọc dữ liệu cleaned_vacdata.rds và vẽ bản đồ thể hiện tỷ lệ tiêm chủng đầy đủ theo từng quận huyện
Tính tỷ lệ trẻ được tiêm ít nhất 3 mũi VGB
Đọc và xử lý dữ liệu
vaccdata <- readRDS("data/cleaned_vacdata.rds")
prop_vaccinated <- vaccdata %>%
group_by(huyen) %>%
summarize(
# Tính số trẻ được tiêm vgb_3 tại mỗi quận
no_vaccinated = sum(khangnguyen == "vgb_3"),
# đếm số trẻ tại mỗi quận
total = n_distinct(id)
) %>%
mutate(
prop_vaccinated = no_vaccinated/total
)Chuẩn bị dữ liệu để vẽ
Code
# ----- đọc bản đồ vn cấp quận huyện ------
vn_qh <- st_read(dsn = "data/gadm41_VNM.gpkg", layer = "ADM_ADM_2")Reading layer `ADM_ADM_2' from data source
`/Users/anhptq/Documents/r4babies/data/gadm41_VNM.gpkg' using driver `GPKG'
Simple feature collection with 710 features and 13 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 102.1446 ymin: 8.381355 xmax: 109.4692 ymax: 23.39269
Geodetic CRS: WGS 84Code
# ----- lọc bản đồ cho hcm ------
hcm_map <- vn_qh %>% filter( NAME_1 == "Hồ Chí Minh" )
# ----- tạo bản đồ thành phố Thủ Đức ------
thuduc_subset <- (hcm_map$VARNAME_2 == "District 2" | hcm_map$VARNAME_2 == "District 9" |
hcm_map$VARNAME_2 == "Thu Duc" )
hcm_map[hcm_map$VARNAME_2 == "Thu Duc", "geom"] <- st_union( subset(hcm_map, thuduc_subset)$geom )
hcm_map <- hcm_map %>% filter(!VARNAME_2 == "District 2" & !VARNAME_2 == "District 9")
# ----- thêm tỷ lệ tiêm chủng ------
hcm_map <- hcm_map %>%
mutate(join_key = tolower(NAME_2)) %>%
left_join(
prop_vaccinated %>% mutate(join_key = tolower(huyen)),
by = join_by(join_key == join_key)
)Cách 1: vẽ bằng ggplot
Code bằng ggplot
hcm_map %>%
ggplot() +
geom_sf(
aes(fill = prop_vaccinated)
) +
scale_fill_binned(
low = "#e5f5e0", high = "#31a354", n.breaks = 8
) +
labs(
title = "Tỷ lệ tiêm VGB mũi 3 theo quận",
fill = "Tỷ lệ tiêm"
)
Cách 2: vẽ bằng leaflet
Code bằng leaflet
# ----- quy định bảng màu ----
pal <- colorBin(
palette = c("#e5f5e0", "#31a354"),
domain = hcm_map$prop_vaccinated,
na.color = "grey"
)
# ----- vẽ biểu đồ ------
leaflet(hcm_map) %>%
# vẽ biểu đồ
addPolygons(
color="black", fillColor = ~pal(prop_vaccinated),
weight = 1, label = ~paste0(NAME_2, ". \nTỷ lệ tiêm: ",
format(prop_vaccinated, digits = 4)),
fillOpacity=1
) %>%
# thêm ô chú thích
addLegend(pal = pal, values = ~prop_vaccinated, title = "Tỷ lệ tiêm", position = "bottomright", opacity=1)